
Foto de grupo. De izquierda a derecha: Iván Butrón Ollo, Diego Vizcaíno Sáez, José Muñoz Dorado, Silvia María Conde Sánchez, Gisela García Ruiz, Juana Pérez Torres, Andrea Pradillo López, Francisco Javier Marcos Torres, Aurelio Moraleda Muñoz
El Grupo Desarrollo Procariótico BIO-318 forma parte del Departamento de Microbiología de la Universidad de Granada (Página web: https://t.co/CnNJBRNFqa; Cuenta en X: https://x.com/MyxoUGR; Cuenta en Instagram: https://www.instagram.com/myxo_ugr/). Este grupo está liderado por el Dr. José Muñoz-Dorado y está formado en la actualidad por dos Catedráticos de Universidad (José Muñoz Dorado y Juana Pérez Torres), un Profesor Titular de Universidad (Aurelio Moraleda Muñoz), un Profesor Permanente Laboral (Francisco Javier Marcos Torres) y diferentes estudiantes de máster y de grado.
El grupo, distinguido con el “Premio Universidad de Granada a la Investigación de Excelencia” en el año 2011, lleva a cabo investigaciones con la mixobacteria modelo Myxococcus xanthus, la cual presenta un comportamiento social y multicelular único entre los procariotas. En nuestros estudios combinamos técnicas clásicas de microbiología, biología molecular y microscopía, con tecnologías de secuenciación masiva, transcriptómica, bioinformática y genómica comparada.
Las mixobacterias: modelo de señalización y multicelularidad procariota
Las mixobacterias (phylum Myxococcota), en general, y M. xanthus, en particular, son depredadores microbianos del suelo que presentan un ciclo celular multicelular peculiar. Así, en presencia de nutrientes, las células se desplazan por deslizamiento de manera coordinada formando enjambres. Cuando los enjambres entran en contacto con la presa, las mixobacterias penetran en la colonia y producen la lisis de sus presas mediante diversos mecanismos, incluyendo la liberación de enzimas hidrolíticas y metabolitos secundarios. Sin embargo, en condiciones de escasez de nutrientes, las mixobacterias, moviéndose coordinada y colectivamente, inician un programa de desarrollo e intercambian señales químicas extracelulares, así como señales dependientes del contacto físico, para formar agregados y construir cuerpos fructíferos de tamaño macroscópico. Durante el ciclo de desarrollo hay tres destinos celulares posibles: i) las células localizadas dentro de los cuerpos fructíferos se diferencian en mixósporas esféricas, que son resistentes a varias condiciones adversas; ii) rodeando el cuerpo fructífero aparece una monocapa de bacilos periféricos, no diferenciados y que permanecen en un estado persistente; y iii) una porción mayoritaria de las células del enjambre experimenta autolisis mediante muerte celular programada. Las mixósporas aseguran la supervivencia durante la inanición o la desecación, y son capaces de dispersarse a otros ambientes y germinar cuando las condiciones nutritivas mejoran (Figura 1A). Tanto la depredación como el ciclo de desarrollo son procesos que requieren la acción coordinada de multitud de células, poniendo de manifiesto la riqueza de sistemas de señalización presentes en estas bacterias para percibir y comunicar rápidamente los cambios en el medio ambiente y coordinar una respuesta grupal.
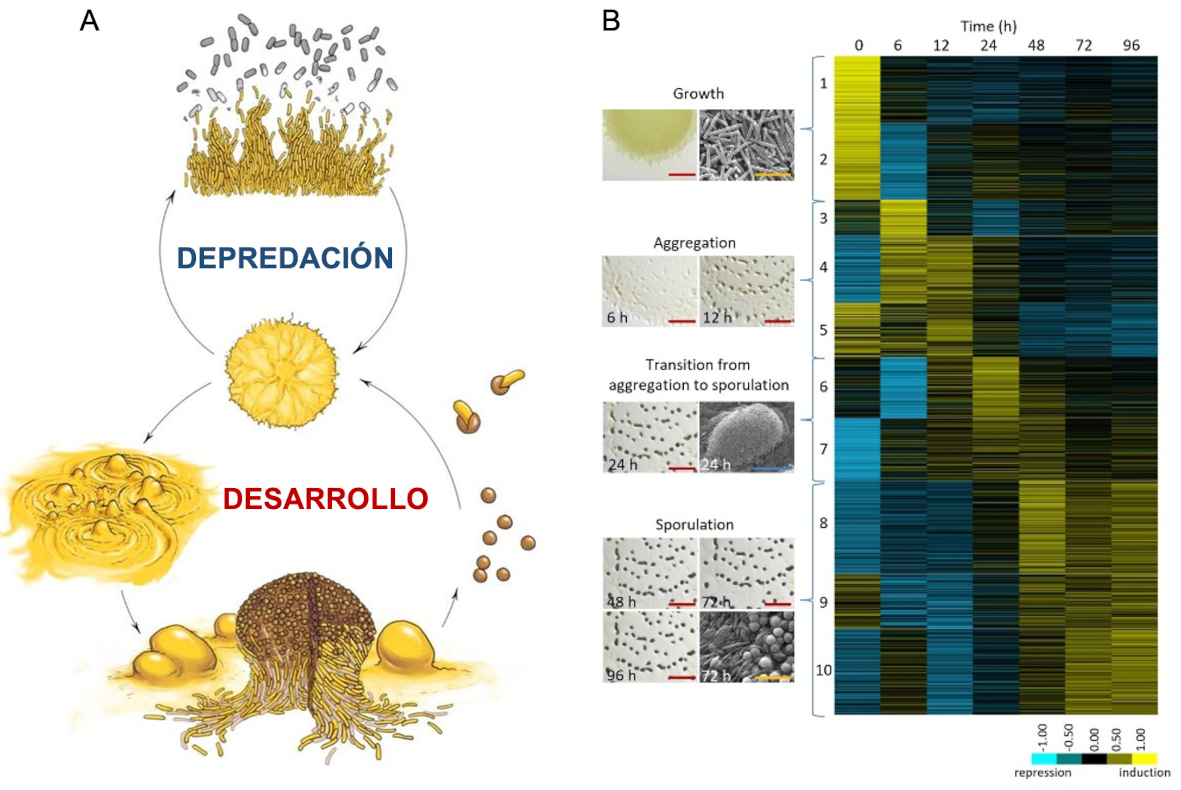
Figura 1. Multicelularidad y señalización en M. xanthus. A. Ciclo de vida de M. xanthus destacando su fase de crecimiento vegetativo donde tiene lugar la depredación cooperativa de otros microorganismos y el ciclo de desarrollo donde se formarán cuerpos fructíferos con mixósporas resistentes a las condiciones adversas. B. Cambios transcriptómicos durante el ciclo de desarrollo de M. xanthus.
Nuestro interés es desentrañar los procesos moleculares que gobiernan los complejos comportamientos sociales de M. xanthus para optimizar su aprovechamiento de los recursos y su supervivencia mediante la adopción de un estilo de vida multicelular, de manera análoga a los organismos eucariotas. En este sentido, nuestros estudios sobre el ciclo de vida de M. xanthus han permitido descifrar la dinámica del transcriptoma a lo largo del ciclo de desarrollo (Figura 1B) y de la depredación (Figura 2A). Asimismo, también estamos interesados a la respuesta de esta bacteria a diversos estímulos de su ambiente. En la actualidad, nuestras dos vías de investigación principales se centran en identificar y caracterizar los mecanismos moleculares que actúan durante la depredación bacteriana y durante la adaptación de esta bacteria a cambios medioambientales.
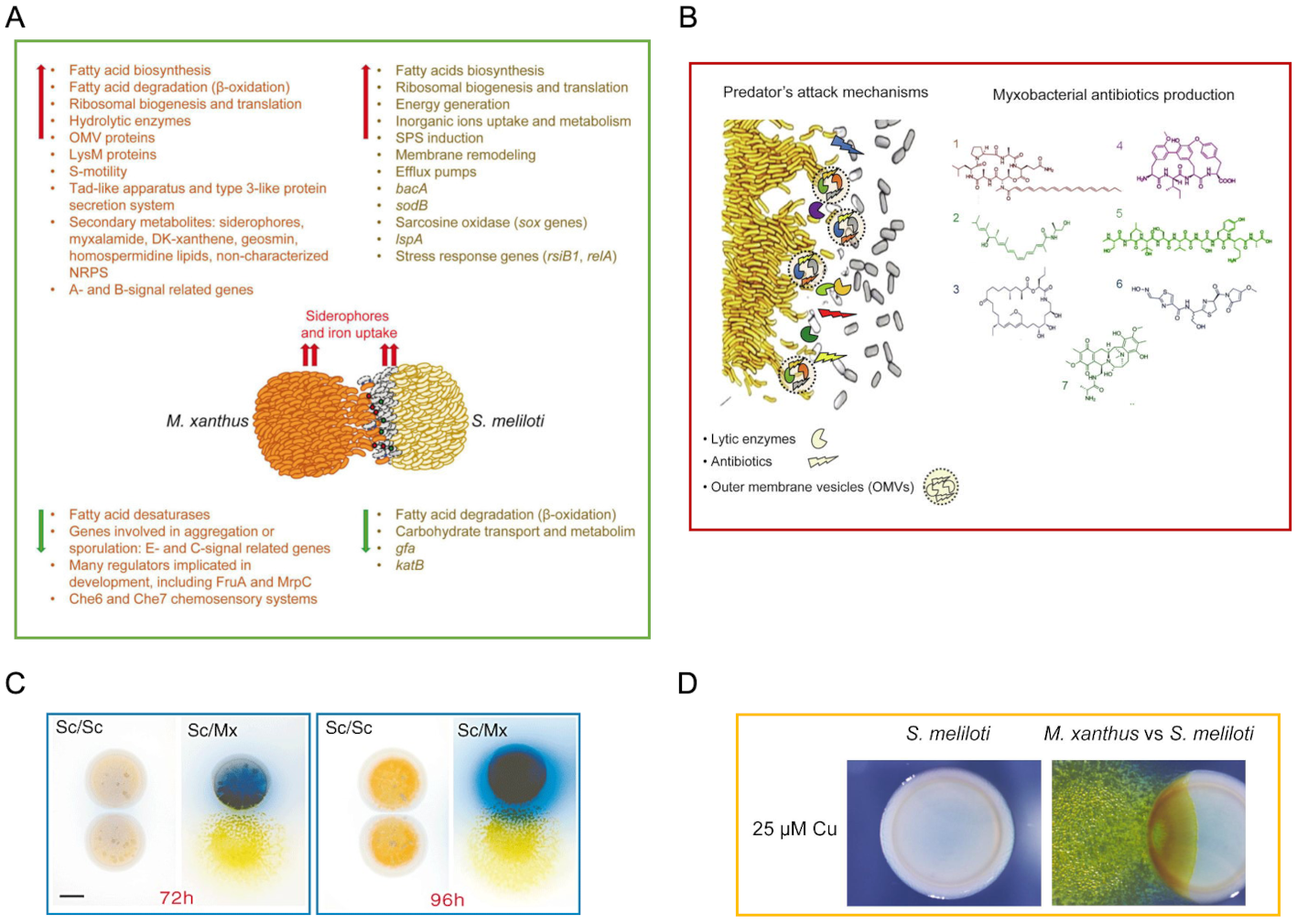
Figura 2. Depredación bacteriana en M. xanthus. A. Análisis del predatosoma de M. xanthus y el defensoma de S. meliloti durante su interacción. B. Producción de antibióticos por parte de M. xanthus durante la depredación. C. Producción de actinorrodina por parte de S. coelicolor cuando es depredada por M. xanthus. D. Producción de melanina por parte de S. meliloti cuando es depredada por M. xanthus en presencia de cobre.
Depredación bacteriana: estudios básicos y consecuencias ecológicas, evolutivas, biotecnológicas y agrícolas
La depredación bacteriana ha sido propuesta como una fuerza impulsora de la evolución. Además, la estructura y composición de las comunidades bacterianas depredadoras comienzan a reconocerse como un factor importante en la biodiversidad, debido a su papel potencial en el control y modelado de las poblaciones de otros microorganismos en el medio ambiente.
- xanthus es capaz de depredar una gran variedad de bacterias Gram positivas y negativas, así como hongos. Nuestro grupo estudia las estrategias de depredación de M. xanthus sobre otras bacterias relevantes del suelo, como Streptomyces coelicolor o Sinorhizobium meliloti. En relación con ello, hemos determinado el predatosoma (genes de M. xanthus implicados en la depredación) y el defensoma (genes de la presa implicados en la defensa) durante la interacción M. xanthus – S. meliloti mediante técnicas de secuenciación masiva (RNA-seq), genómica, y técnicas clásicas de biología molecular y microbiología (Figura 2A). Esta investigación ha arrojado luz sobre los procesos utilizados por estos microscópicos depredadores para reconocer, matar y lisar a sus presas.
Por otro lado, dado que las mixobacterias poseen potencial genético para producir una amplia batería de antibióticos y otros metabolitos secundarios (Figura 2B), los estudios in vivo frente a presas pueden contribuir al descubrimiento de nuevos productos con actividad biológica y podrían abrir nuevos horizontes en su aplicación a procesos de biocontrol contra bacterias patógenas, como alternativa a los antibióticos.
Además, el estudio de los mecanismos de defensa de la presa nos está permitiendo desvelar nuevas estrategias bacterianas frente a la depredación, como es la inducción de la producción de nuevos antibióticos en el caso de S. coelicolor (Figura 2C) o metabolitos de protección contra el estrés oxidativo, como en el caso de Sinorhizobium, que produce melanina para protegerse de M. xanthus cuando es depredado en presencia de cobre (Figura 2D).
Adaptación de M. xanthus a cambios medioambientales
- xanthus debe completar su ciclo de vida en presencia de compuestos naturales propios de su entorno. Uno de estos compuestos es el cobre, que se encuentra en concentraciones que pueden oscilar entre 2 y 100 mg/kg de suelo. Este metal es esencial para la vida, ya que actúa como cofactor de numerosas enzimas implicadas en procesos vitales pero, sin embargo, si se encuentra en exceso, también resulta extremadamente tóxico, principalmente por la formación de especies reactivas de oxígeno. Debido a este doble efecto, las células poseen distintos mecanismos para asegurar niveles intracelulares adecuados de cobre. M. xanthus nos ofrece la oportunidad de estudiar la respuesta celular a lo largo de un ciclo de vida completo, lo que nos ha permitido dilucidar la respuesta global al cobre en monocultivos y en presencia de presa (Figura 3A). Además, estos estudios nos han dado la posibilidad de identificar varios componentes de la respuesta al cobre que podrían estar implicados en la resistencia cruzada a antibióticos. El estudio de la regulación de los genes implicados en la homeostasis del cobre también nos ha permitido describir nuevos sistemas de regulación, incluyendo un nuevo grupo de factores sigma ECF dependientes de metales que, debido a su capacidad de unir metales y a su mecanismo de acción independiente de factores antisigma, ha supuesto la creación de un nuevo grupo de metalorreguladores (Figura 3B).
Además del cobre, nuestro grupo también ha estudiado los efectos sobre M. xanthus de otros elementos presentes en el medio ambiente como pueden ser la luz, el cadmio, el zinc o el hierro. Acerca de este último, recientemente hemos descrito que juega un papel clave durante la depredación influyendo en el resultado de dicha interacción (Figura 3C).
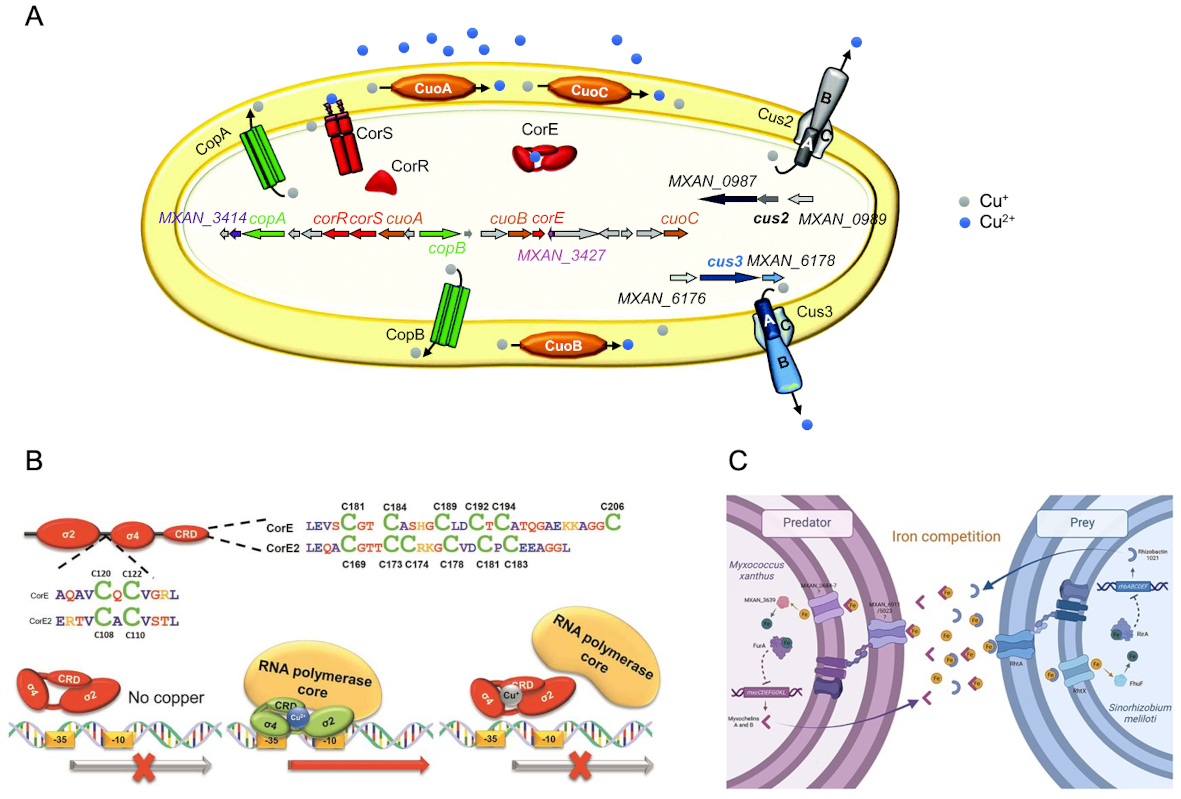
Figura 3. Adaptación a cambios medioambientales en M. xanthus. A. Respuesta global al cobre en M. xanthus. B. Mecanismo de regulación de los factores sigma de tipo ECF CorE-like. C. Papel del hierro durante la depredación bacteriana de M. xanthus.
TRABAJOS SELECCIONADOS (ÚLTIMOS 10 AÑOS)
Kroos, L., Wall, D., Islam, S.T., Whitworth, D.E., Muñoz-Dorado, J., Higg, P.I., Singer, M., Mauriello, E.M. , et al. (2025). Milestones in the development of Myxococcus xanthus as a model multicellular bacterium. Journal of Bacteriology, 207:e0007125
Contreras-Moreno, F.J., Moraleda-Muñoz. A., Marcos-Torres, F.J., Cuéllar, V., Soto, M.J., Pérez, J. and Muñoz-Dorado, J. (2024). Siderophores and competition for iron govern myxobacterial predation dynamics. ISME Journal, 2:wrae077.
Contreras-Moreno, F.J., Pérez, J., Muñoz-Dorado, J., Moraleda-Muñoz, A. and Marcos-Torres, F.J. (2024). Myxococcus xanthus predation: an updated overview. Frontiers in Microbiology, 15: 1339696
Soto, M.J., Pérez, J., Muñoz-Dorado, J., Contreras-Moreno, F.J., and Moraleda-Muñoz, A. (2023). Transcriptomic response of Sinorhizobium meliloti to the predatory attack of Myxococcus xanthus. Frontiers Microbiology 14:1213659.
Marcos-Torres, F.J., Moraleda-Muñoz, A., Contreras-Moreno, F.J., Muñoz-Dorado, J. and Pérez, J. (2022). Mechanisms of action of non-canonical ECF sigma factors. International Journal of Molecular Sciences, 23: 3601-3614
Pérez, J., Contreras-Moreno, F.J., Muñoz-Dorado, J., Moraleda-Muñoz, A. (2022). Development versus predation: Transcriptomic changes during the lifecycle of Myxococcus xanthus. Frontiers Microbiology 13: 1004476
Li, Y.P., Ben-Fekih, I., Chi-Fru, E., Moraleda-Muñoz, A., Li, X., Rosen, B.P., Yoshinaga, M. y Rensing, C. (2021). Antimicrobial activity of metals and metalloids. Annual Review of Microbiology, 75:1, 175-197
Marcos-Torres, F.J., Volz, C., and Müller R. (2020). An ambruticin-sensing complex modulates Myxococcus xanthus development and mediates myxobacterial interspecies communication. Nature Communications, 11: 5563.
Contreras-Moreno, F.J., Muñoz-Dorado, J., García-Tomsig, N.I., Martínez-Navajas, G., Pérez, J., and Moraleda-Muñoz. (2020). Copper and melanin play a role in Myxococcus xanthus predation on Sinorhizobium meliloti. Frontiers in Microbiology, 11: 94.
Pérez, J., Contreras-Moreno, F.J., Marcos-Torres, F.J., Moraleda-Muñoz, A. and Muñoz-Dorado. J. (2020). The antibiotic crisis: How bacterial predators can help. Computational and Structural Biotechnology Journal, 118: 2547–2555.
Muñoz-Dorado J., Moraleda-Muñoz, A., Marcos-Torres, F.J., Contreras-Moreno, F.J., Martin-Cuadrado, A.B., Schrader, J.M., Higgs, P.I., and Pérez, J. (2019). Transcriptome dynamics of the Myxococcus xanthus multicelular developmental program. eLife: e50374.
Moraleda-Muñoz, A., Marcos-Torres, F.J., Pérez, J., and Muñoz-Dorado J. (2019). Metal-responsive RNA polymerase extracytoplasmic function (EFC) sigma factors. Molecular Microbiology 112: 385-398.
Pérez, J., Muñoz-Dorado J., and Moraleda-Muñoz, A. (2018). The complex global response to copper in the multicellular bacterium Myxococcus xanthus. Metallomics. 10: 876-886.
Muñoz-Dorado J., Marcos-Torres, F.J., Moraleda-Muñoz, A., and Pérez J. (2016). Myxobacteria: Moving, killing, feeding, and surviving together. Frontiers in Microbiology. 4: 1-18.
Marcos-Torres, F.J., Pérez, J., Gómez-Santos, N., Moraleda-Muñoz, A., and Muñoz-Dorado, J. (2016). In depth analysis of the mechanism of action of metal-dependent sigma factors: characterization of CorE2 from Myxococcus xanthus. Nucleis Acids Research. 44: 5571-5584.
Pérez, J., Moraleda-Muñoz, A., Marcos-Torres, F.J., and Muñoz-Dorado, J. (2016). Bacterial predation: 75 years and counting!. Environmental Microbiology. 18: 766-779.
