
Foto de grupo. De izquierda a derecha. Sol Vendrell, Leticia Lucero, Cristina Fernández, Rafael Giraldo, Silvia Marín
En nuestro grupo de investigación abordamos un programa de Biología Sintética (BS) cuyo objetivo es el desarrollo de piezas, dispositivos y sistemas para el modelado de proteinopatías amiloides en microorganismos (Giraldo, 2010). Pretendemos así el deconstruir las amiloidosis en módulos funcionales cuya integración haga posible su mejor comprensión y también, potencialmente, actuaciones con finalidad diagnóstica y/o terapéutica. Las amiloidosis son procesos degenerativos, neuronales o sistémicos, de una extraordinaria complejidad desde los puntos de vista molecular, celular y de organismos completos (Eisenberg y Jucker, 2012). Sin embargo, en todos los casos estudiados, el agente causal de cada tipo de amiloidosis es relativamente simple: una proteína, como Aβ y Tau (en la enfermedad de Alzheimer), α-sinucleína (en Parkinson), Htt (en Huntington) o SOD1 (en ALS), que presenta al menos dos conformaciones; mientras que la nativa es soluble, la otra alternativa agrega formando fibras con un esqueleto β-laminar amiloide. Estos agregados amiloides, más específicamente sus oligómeros precursores o los resultantes de un desensamblaje parcial, son las especies moleculares citotóxicas. Una propiedad fascinante de toda proteína amiloide es la capacidad de replicar su conformación aberrante al moldearla por contacto en moléculas solubles de esa misma proteína. En realidad, la forma ensamblada está compuesta por un conjunto de conformaciones metaestables cuyas energías libres son muy similares, estando por ello en equilibrio las unas con las otras. Esto se manifiesta, para una proteína determinada, en la existencia de «estirpes» o variantes conformacionales y de «cuasiespecies», v.g., familias de conformaciones relacionadas sometidas a selección y replicación (Giraldo, 2010; Eisenberg y Jucker, 2012). Las proteínas amiloides guardan una relación estrecha con los priones, al ser éstos una subclase de amiloides pero con una propiedad adicional que nos es bien conocida como microbiólogos: la infectividad, es decir, su transmisibilidad «horizontal», siquiera en condiciones experimentales, entre células u organismos completos dentro de una población (Eisenberg y Jucker, 2012). El prion arquetípico es PrP, cuyas diversas conformaciones agregadas amiloides están detrás de las encefalopatías espongiformes transmisibles en mamíferos, mientras que los agentes de las demás amiloidosis son denominados «prionoides» al carecer de infectividad. Sin embargo, los estudios más recientes parecen concluir que en todos los procesos neurodegenerativos las conformaciones amiloides siguen, en su dispersión progresiva por el sistema nervioso, una propagación intercelular de tipo infectivo, lo que permitiría calificarlos también, sensu lato, como priones (Prusiner, 2012).
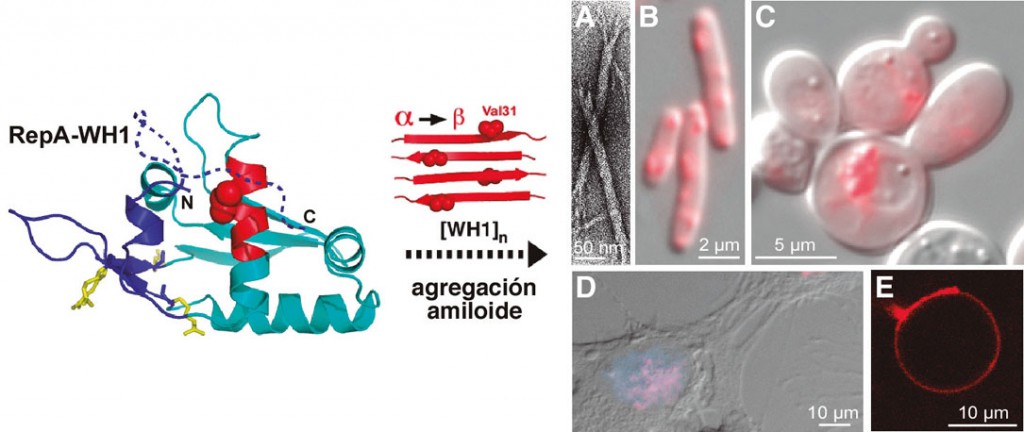
Figura 1. El dominio WH1 de la proteína de replicación plasmídica RepA (izda.), al monomerizar cambia parte de su estructura a-helicoidal en hebras-b (en rojo) y se ensambla como fibras amiloides (A, micrografía electrónica). Al fusionarla a la proteína fluorescente mCherry, RepA-WH1 aparece como inclusiones en el citoplasma de E. coli (B) y S. cerevisiae (C), microorganismos en los que actúa como prionoide, así como al expresarla en el núcleo (azul) de células de neuroblastoma en cultivo (D). En liposomascitomiméticos, RepA-WH1 agrega en la bicapa lipídica (E).
La BS de módulos amiloides proteotóxicos en microorganismos como la bacteria Gram-negativa Escherichia coli o la levadura Saccharomyces cerevisiae es un reto formidable, pues a la complejidad propia de la bioingeniería de proteínas se une el que las bacterias no sufren, de manera natural, amiloidosis intracelulares, mientras que en las levaduras las amiloidosis no son citotóxicas, sino un recurso funcional que confiere adaptaciones fenotípicas rápidas y transmisibles epigenéticamente (Blanco et al., 2012). Sin embargo, por esas mismas razones, la BS nos ofrece la oportunidad de desarrollar modelos microbianos de amiloidosis en los que tanto los agentes etiológicos como sus células hospedadoras pueden ser concebidos ab nitio como módulos ortogonales, es decir, sin interacción funcional previa (Giraldo, 2010). Al establecerse nuestro grupo en 1999 nos interesaba el estudiar la estructura y el mecanismo de acción de las proteínas, denominadas Rep, que inician la replicación del DNA de los plásmidos bacterianos y, de manera colateral, el ensamblaje de las proteínas del complejo de replicación ORC en S. cerevisiae (Giraldo, 2003). Descubrimos que la proteína RepA codificada por el plásmido pPS10 experimenta un cambio conformacional que la faculta para pasar de ser un dímero, represor transcripcional del propio gen repA, a ser un monómero iniciador de la replicación, proceso activado por la unión a secuencias de DNA específicas que conlleva una remodelación estructural del dominio N-terminal de la proteína (WH1) (Giraldo et al, 2003). RepA-WH1 incrementa así su estructura b-laminar y, finalmente, la proteína agrega en un complejo postreplicativo que inhibe reinicios prematuros de la replicación (Gasset-Rosa et al, 2008a). En 2007 determinamos que RepA-WH1 se ensambla in vitro en fibras amiloides en un proceso promovido por su interacción con oligodesoxirribonucleótidos (Giraldo, 2007; Gasset-Rosa et al, 2008b), lo que nos llevó a explorar si su expresión en la bacteria E. coli, desacoplada de la replicación plasmídica, conducía a la formación de agregados amiloides intracelulares. Efectivamente, RepA-WH1, fusionada a una proteína reportera fluorescente, forma inclusiones citoplasmáticas que resultan citotóxicas para las bacterias y que, una vez purificadas, son capaces de acelerar invitro la fibrilación amiloide (Fernández-Tresguerres et al., 2010). RepA-WH1, dada la ausencia de proteínas homólogas en el proteoma humano en las que pudiera moldear su conformación amiloide y su carácter no infectivo, es clasificable como un prionoide con nivel de bioseguridad 1. Recientemente, en colaboración con el grupo de A. Lindner (INSERM – U. Descartes, París) y gracias en parte a una beca de la FEMS, hemos estudiado la transmisión «vertical» (de célula madre a células hijas) de los agregados de RepAWH1 mediante técnicas de microfluídica. Dichos estudios nos han llevado a caracterizar la existencia de dos estirpes amiloides, fenotípicamente diferenciadas por su toxicidad y la morfología de los agregados, cuya interconversión es dependiente de un único factor celular: DnaK, la chaperona de la familia Hsp70 en E. coli (Gasset-Rosa et al., 2014; Molina-García y Giraldo, 2014). Aproximaciones transcriptómicas y proteómicas aún en curso nos están permitiendo el trazar, con una nitidez difícilmente alcanzable en sistemas celulares complejos, las rutas de citotoxicidad amiloide. Entre las dianas comunes a las proteínas implicadas en las amiloidosis humanas destaca la membrana plasmática, por lo que estamos estudiando también la interacción de RepA-WH1 con liposomas, llevando así la modelización de dichos procesos al extremo del minimalismo citomimético. En dirección diametralmente opuesta, explorando sistemas más complejos, hemos podido establecer (Gasset-Rosa y Giraldo, 2014) que la secuencia responsable de la amiloidosis en RepA-WH1, si se dispone en forma de repeticiones en tándem, es capaz de sustituir en quimeras a las propias del prion de levaduras Sup35p/[PSI+] (Liebman y Chernoff, 2012), generando así un nuevo prión sintético funcional, que hemos denominado [REP-PSI+]. Éste se manifiesta en forma de diversas variantes fenotípicas cuya interconversión está regulada, al igual que en su parental RepA-WH1, por una chaperona Hsp70 (Ssa1p) y no por Hsp104p, la chaperona que modula la propagación de [PSI+] en las levaduras (Liebman y Chernoff, 2012). Por último, estamos explorando la expresión de RepA-WH1 en células humanas en cultivo y desarrollamos sistemas para el cribado en bacterias de moléculas inhibidoras de la amiloidosis, que constituirán pruebas de concepto del potencial de los sistemas bacterianos sintéticos en la modelización y el abordaje farmacológico de proteinopatías amiloides con relevancia biomédica.
Información actualizada del grupo puede encontrarse en:
Bibliografía representativa
Blanco LP, Evans ML, Smith DR, Badtke MP y Chapman MR. (2012). Diversity, biogenesis and function of microbial amyloids. Trends Microbiol 20: 66–73.
Eisenberg D y Jucker M. (2012). The amyloid state of proteins in human diseases. Cell 148: 1188–1203.
Fernández-Tresguerres ME, Moreno-Díaz de la Espina S, Gasset-Rosa F y Giraldo R. (2010). A DNA-promoted amyloid proteinopathy in Escherichia coli. Mol Microbiol 77: 1456–1469.
Giraldo R. (2003). Common domains in the initiators of DNA replication in Bacteria, Archaea and Eukarya: Combined structural, functional and phylogenetic perspectives. FEMS Microbiol Rev 26: 533–554.
Giraldo R. (2007). Defined DNA sequences promote the assembly of a bacterial protein into distinct amyloid nanostructures. Proc Natl Acad Sci USA 104: 17388–17393.
Giraldo R. (2010). Amyloid assemblies: Protein legos at a crossroads in bottom-up Synthetic Biology. ChemBioChem 11: 2347–2357.
Giraldo R, Fernández-Tornero C, Evans PR, Díaz-Orejas R y Romero A. (2003). A conformational switch between transcriptional repression and replication initiation in the RepA dimerization domain. Nat Struct Biol 10: 565–571.
Gasset-Rosa F, Díaz-López T, Lurz R, Prieto A, Fernández-Tresguerres ME y Giraldo R. (2008a). Negative regulation of pPS10 plasmid replication: Origin pairing by zipping-up DNA-bound RepA monomers. Mol Microbiol 68: 560–572.
Gasset-Rosa F, Maté MJ, Dávila-Fajardo C, Bravo J, y Giraldo R. (2008b). Binding of sulphonated indigo derivatives to RepA-WH1 inhibits DNA-induced protein amyloidogenesis Nucleic Acids Res 36: 2249–2256.
Gasset-Rosa F, Coquel AS, Moreno-del Álamo M, Chen P, Song X, Serrano AM, Fernández-Tresguerres ME, Moreno-Díaz de la Espina S, Lindner AB y Giraldo R. (2014). Direct assessment in bacteria of prionoid propagation and phenotype selection by Hsp70 chaperone. Mol Microbiol 91: 1070–1087.
Gasset-Rosa F y Giraldo R. (2014). The Hsp70 chaperone Ssa1p selects for weak variants of [REP-PSI+], a novel bacterial-yeast hybrid prion. En revisión.
Liebman SW y Chernoff YO. (2012). Prions in yeast. Genetics 191:1041–1072.
Molina-García L y Giraldo R. (2014). Aggregation interplay between variants of the RepA-WH1 prionoid in Escherichia coli. J Bacteriol 196: 2536–2542.
Prusiner SB. (2012). A unifying role for prions in neurodegenerative diseases. Science 336
