
Foto de grupo. De izquierda a derecha: Montserrat Argandoña, Joaquín J. Nieto, Francine Piubeli, Carmen Vargas, Rosa García, Emilia Naranjo, Alí Tahrioui y Manuel Salvador.
Nuestro Grupo de investigación (Grupo BIO-320 de la Consejería de Economía, Innovación, Ciencia y Empleo de la Junta de Andalucía) tiene por objeto el estudio de la genética molecular de los procesos de osmoadaptación en procariotas, en concreto en un grupo de microorganismos extremófilos, las bacterias halófilas moderadas, y su aplicación en la producción de compuestos bioestabilizadores útiles en la industria Farmacéutica, Agricultura, y en Biomedicina. Se trata de un Grupo, liderado por los Profs. Carmen Vargas Macías y Joaquín J. Nieto, constituido por 7 doctores y dos graduados, que posee una amplia experiencia en esta línea de investigación, subvencionada en los últimos 20 años por numerosos proyectos nacionales e internacionales del Plan Nacional, de la Junta de Andalucía y de la Comisión Europea.
Chromohalobacter salexigens, es una gamma-proteobacteria que está considerada como un excelente modelo biológico para estudiar los procesos de osmoadaptación en bacterias, ya que posee uno de los rangos salinos de crecimiento más amplios de los conocidos (0,5 a 3 M de NaCl en medio mínimo). Para poder compensar este estrés osmótico, es capaz de acumular intracelularmente grandes cantidades de solutos compatibles, compuestos polares de bajo peso molecular, ya sea tras su transporte desde el medio externo si están presentes, como es el caso de la betaína, o mediante su síntesis, en ausencia de dichos compuestos en el medio. Esta estrategia es muy versátil, ya que le permite adaptarse a muy diferentes salinidades externas ajustando la concentración intracelular de dichos solutos compatibles, ya que puede también expulsarlos al exterior si la presión osmótica disminuye en el medio externo. Los compuestos que sintetiza mayoritariamente en respuesta a un estrés osmótico y térmico son las ectoínas (ectoína e hidroxiectoína), unos diaminoácidos que han adquirido en los últimos años un gran interés industrial. En efecto, estos compuestos, que sólo se pueden obtener mediante síntesis biológica, poseen excelentes propiedades bioestabilizadoras y protectoras, por lo que se utilizan en Dermofarmacia como componentes de geles y cremas antiedad, cremas de protección frente rayos UV, etc.., y en Biología Molecular como protectores de anticuerpos, enzimas, ácidos nucléicos, entre otros, presentando además un enorme potencial en el campo de la Biomedicina. Así, se ha descrito recientemente que inhiben in vitro la formación de los agregados de betaamiloide característicos de las enfermedades neurodegenerativas como el Alzheimer. Dado que las ectoínas se acumulan sobre todo a elevada salinidad y temperatura, C. salexigens es un excelente ejemplo de factoría celular para la producción de las mismas. Entre las ventajas que presenta se encuentra la facilidad para su cultivo usando medios convencionales, su gran versatilidad metabólica, la disponibilidad de herramientas genéticas para su manipulación (la mayoría desarrolladas por nuestro Grupo) y, muy importante, el hecho de se disponga de la secuencia completa de su genoma.
En los últimos años, hemos realizado numerosos estudios encaminados a dilucidar los mecanismos moleculares de osmoadaptación en C. salexigens. Así, hemos demostrado que, además de ciertas modificaciones en la composición lipídica de sus membranas, esta bacteria es también capaz de sintetizar trehalosa implicada en la protección frente a elevada temperatura y desecación, mediante los genes otsAB, así como betaína, a partir de su precursor, la colina, mediante los genes betIBA. También hemos caracterizado a nivel molecular los genes de la síntesis de ectoína (ectABC y su derivado hidroxilado, la hidroxiectoína (ectD, ectE), así como los genes que codifican los sistemas de captación de las mismas desde el medio externo (teaABC). La síntesis de ectoínas en C. salexigens así como su regulación, es extraordinariamente compleja, estando osmo- y termorregulada, y en la que intervienen diferentes reguladores (sS, s32, Fur, etc), habiendo demostrado por primera vez en bacterias halófilas la interrelación entre la homeostasis de hierro y la osmoadaptación
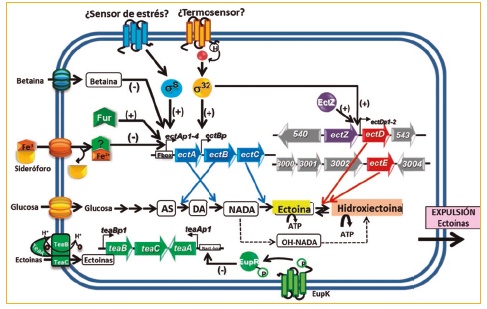
Figura 1. Esquema de los mecanismos de osmoadaptación de C. salexigens
Recientemente hemos caracterizado a nivel molecular un regulador de respuesta (EupR) de un sistema de dos componentes implicado en la osmodetección, el primero descrito para bacterias halófilas, que parece mediar en el control transcripcional del sistema de captación de ectoínas, codificado por los genes teaABC. Además existen rutas de síntesis alternativas y de degradación, actualmente en estudio (Figura 1). Todo ello dificulta en gran medida la obtención de cepas mutantes productoras de ectoínas que sean capaces de superproducir estos compuestos en condiciones más favorables a nivel industrial que las actuales, como es, por ejemplo, a temperatura ambiente o en condiciones de baja salinidad.Para ello, nuestro Grupo aborda este objetivo en los últimos años mediante la aproximación de la Biología de Sistemas, en la que se conjugan los datos provenientes de la aplicación de técnicas de Biología Molecular de última frontera,como son la proteómica,la transcriptómica, la metabolómica y la flujómica, con métodos bioinformáticos de modelado in silico (Fig. 2).
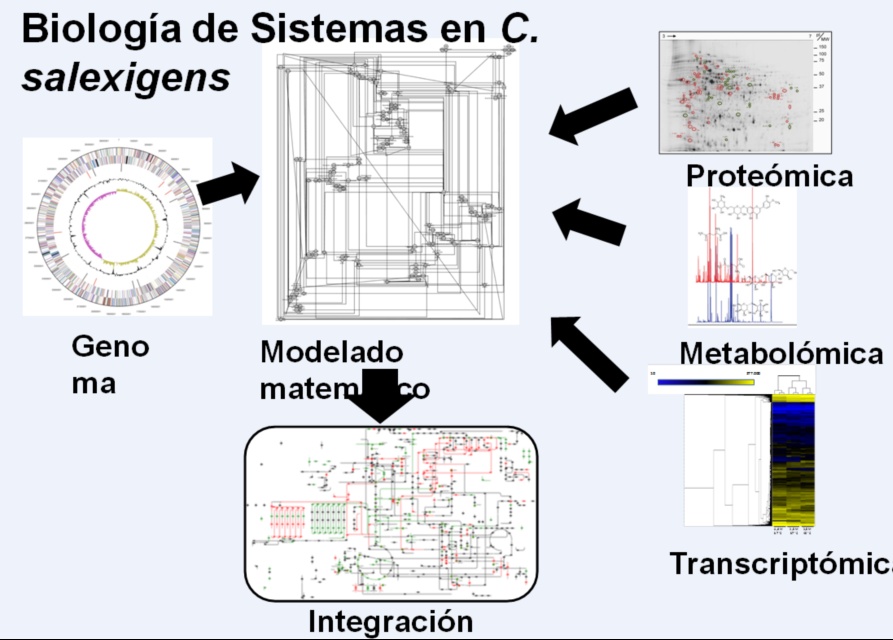
Figura 2. Biología de Sistemas de C. salexigens
A este respecto, hemos completado la primera reconstrucción metabólica refinada a escala genómica de C. salexigens, en la que se ha conectado el metabolismo del C y el N con todas las rutas relativas al transporte, síntesis, degradación y eflujo de los principales solutos compatibles acumulados por esta bacteria.Esta red metabólica se ha utilizado para obtener un primer modelometabólico matemático que refleja con bastante precisión el sistema biológico de estudio,ya que ha sido manualmente revisado en base a estudios bioquímicosy fisiológicos previos, y a datos provenientes de análisis multiómicos recientemente llevados a cabo por nuestro Grupo.Además ha sido validado comparando los resultados de las simulaciones in silico con los datos de resultados experimentales (determinación de las cinéticas de crecimiento con diferentes fuentes de carbono y energía y nitrógeno a diferentes salinidades , el análisis de metabolitos extracelulares, la determinación de actividades enzimáticas del metabolismo central, proteómica diferencial, entre otros).
Toda la información obtenida a partir de este modelo nos ayudará a predecir mejor las respuestas fisiológicas y así desarrollar nuevas estrategias para optimizar la obtención de cepas de C. salexigens hiperproductoras de ectoínas.
Por otro lado, hemos obtenido nuevas cepas mutantes productoras de hidroxiectoína en las que se ha conseguido desacoplar la síntesis de su regulación por temperatura y salinidad, obteniéndose valores de producción de este soluto compatible a 37ºC y 0,75 M NaCl similares a los de la cepa silvestre a 45º C y 2,5 M NaCl, lo que supone una gran ventaja desde el punto de vista industrial (resultados que han sido protegidos bajo patente). Por último, por las razones anteriormente expuestas, estamos muy interesados en el estudio molecular del efecto neuroprotector de las ectoinas y su posible aplicación en Biomedicina, para lo cual el Grupo ya cuenta con la colaboración a nivel de proyectos de investigación subvencionados con diversas empresas del sector interesadas en el tema.
Pastor JM, Bernal V, Salvador M, Argandoña M, Vargas C, Csonka L, Nieto JJ y Cánovas M. (2013). Role ofcentral metabolism in the osmoadaptation of the bacterium C.salexigens. J Biol Chem 288:17769-81.
Rodríguez-Moya J, Argandoña M, Nieto JJ, Iglesias-Guerra F y Vargas C. (2013). Temperature- and salinity-decoupled overproduction of hydroxyectoine by C. salexigens. Appl Environ Microbiol 79: 1018-1023.
Reina-Bueno M, Argandoña M, Salvador M, Csonka L, Nieto JJ y Vargas C. (2012). Role of trehalose intolerance to salinity, temperature and desiccation in C. salexigens. PLoS One 7(3). 2012e33587.
Argandoña M, Vargas C, Reina-Bueno M, Salvador M y Nieto JJ. (2012). An extended suite of genetic tools for use in bacteria of the Halomonadaceae: an overview. In: P Balbas and A Lorence (eds), Methods in Molecular Biology, Reviews and Protocols, 3rd ed, vol. 824, Humana Press, Totowa, NJ, pp. 167-201.
Argandoña M, Nieto JJ, Calderón MI, García-Estepa R y Vargas C. (2011). Interplay between iron homeostasis and the osmostress response in the halophilic C. salexigens. Appl Environ Microbiol 76: 3575-3589.
Copeland A, O’Connor K, et al. (2011). Complete genome sequence of the halophilic and highly halotolerant Chromohalobacter salexigens type strain (1H11T). Stand Genom Sci 5:379-88.
Pastor JM, Salvador M, Argandoña M, Bernal V, Reina-Bueno M, Vargas C, Nieto JJ y Cánovas M. (2010). Ectoines in cell stress protection: uses and biotechnological production. Biotechnol Adv 28: 782-807.
Rodríguez-Moya J, Argandoña M, Reina-Bueno M, Nieto JJ, y Vargas C. (2010). Involvement of a two-component system in the control of the uptake of ectoines by the halophilic bacterium C. salexigens. BMC Microbiol 10: 256-274.
Vargas C, Argandoña M, García-Estepa R, Reina-Bueno M, Rodríguez-Moya J y Nieto JJ. (2008). Unravelling the adaptation responses to osmotic and temperature stress in C. salexigens, a bacterium with broad salinity tolerance. Saline Systems 4: 14–23.
García-Estepa R, Argandoña M, Reina-Bueno M, Iglesias-Guerra F, Nieto JJ y Vargas C. (2006). The ectD gene, which is involved in the synthesis of the compatible solute hydroxyectoine, is essential for thermoprotection of the halophilic bacterium Chromohalobacter salexigens. J Bacteriol 188: 3774 – 84.
