Biología de las interacciones beneficiosas planta-microorganismo
Departamento de Microbiología de la Facultad de Biología de la Universidad de Sevilla. Avda. de Reina Mercedes 6, 41012-Sevilla.
Actualmente, dentro del grupo de investigación hay seis miembros que actúan como investigadores principales y que pertenecen al grupo PAIDI de la Junta de Andalucía, BIO-169: José María Vinardell González, Francisco Javier López Baena, Carlos Medina Morillas, Patricia Bernal Guzmán (> ver más), Jose Manuel Borrero de Acuña (> ver más) y Francisco Pérez Montaño (> ver más).
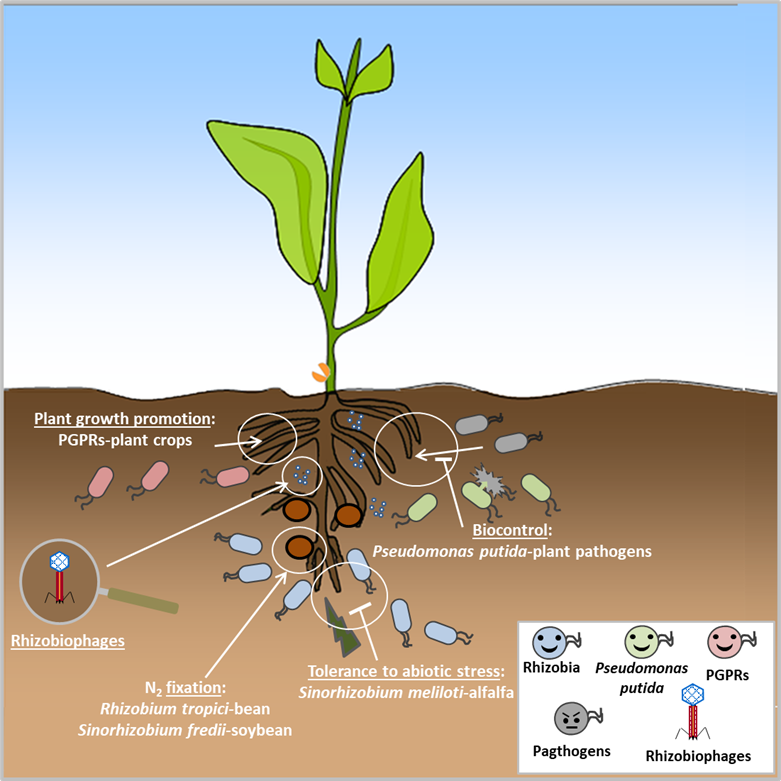
El grupo de investigación estudia diversas rizobacterias que promueven el crecimiento de plantas de interés agrícola mediante biofertilización, bioestimulación y/o biocontrol. Principalmente investigamos las señales moleculares que rigen la interacción simbiótica fijadora de N2 atmosférico establecida entre rizobios y leguminosas (factores de nodulación, polisacáridos de superficie y efectores del sistema de secreción de tipo III o T3SS) y los mecanismos utilizados por bacterias para controlar el crecimiento, la colonización o la infección de bacterias fitopatógenas en cultivos de interés agrícola. Nos centramos especialmente en aquellos agentes de biocontrol que usan para ello el sistema de secreción de tipo VI o T6SS y las vesículas de membrana.
Utilizamos los modelos: a) Sinorhizobium fredii–soja, b) Rhizobium tropici–judía, c) Sinorhizobium meliloti-alfalfa, y d) Pseudomonas putida-fitopatógenos.
Nuestro objetivo es generar conocimiento que contribuya a una agricultura sostenible y a su adaptación a ambientes adversos, con el fin de generar alternativas y así evitar, o al menos disminuir, el uso de fertilizantes y pesticidas químicos.
A continuación se detallan los componentes del grupo PAIDI por orden alfabético:
BIO169: Biotecnología de la Interacción de Microorganismos con leguminosas y otras plantas de interés agrícola
o Azogue Palma, Carlos – Becario contratado de proyecto
o Ayala García, Paula – Becaria predoctoral FPU
o Bernal Guzmán, Patricia – Contratada Ramón y Cajal
o Borrero de Acuña, José Manuel – Contratado Emergia
o Buendía Clavería, Ana María – Profesora Titular de Universidad
o Cubo Sánchez, María Teresa – Profesora Titular de Universidad
o Del Cerro Sánchez, Pablo – Contratado Ramón y Cajal
o Espuny Gómez, María del Rosario – Catedrática de Universidad
o Fuentes Romero, Francisco – Becario predoctoral Junta de Andalucía
o García Rodríguez, Diego – Becario predoctoral PIF
o Herrero Gómez, Irene – Becaria predoctoral FPU
o Jacott, Catherine Nancy – Investigadora Juan de la Cierva formación
o Jiménez Guerrero, Irene – Investigadora Juan de la Cierva incorporación
o López Baena, Francisco Javier – Profesor Titular de Universidad
o Medina Morillas, Carlos – Profesor Titular de Universidad
o Moreno de Castro, Natalia – Becaria predoctoral FPU
o Ollero Márquez, Francisco Javier – Catedrático de Universidad
o Pérez Montaño, Francisco – Profesor Titular de Universidad
o Reyes Pérez, Pedro José – Becario predoctoral contratado de proyecto
o Vinardell González, José María – Catedrático de Universidad
JoseBorrero
Engineering extracellular membrane vesicles from rhizospheric bacteria for the development of biopesticides and plant-growth promoting agents
Ingeniería de vesículas extracelulares de membrana de bacterias rizosféricas para el desarrollo de biopesticidas y agentes promotores del crecimiento vegetal
IP: Jose Manuel Borrero de Acuña
Research in our lab:
One of our major endeavours is to elucidate protein complex assembly dynamics involved in crucial biological processes for diverse bacteria. The majority of cellular functions in all living cells are conducted by proteins. Biochemical pathways, signal transduction cascades, and membrane-associated complexes of energy generation rely on the fine-tuned, directed and frequently transient protein interactions. We have widely investigated biochemical processes for relevant human pathogens (Legionella pneumophila, Clostridioides difficile and P. aeruginosa) and environmentally relevant bacteria (Dinoroseobacter shibae and Pseudomonas putida); (see publications: https://scholar.google.es/citations?user=lo_iLZQAAAAJ&hl=es&oi=ao). Dissecting the protein-protein interactions underlying such processes is crucial for basic research but it is also a cornerstone for boundless biomedicine and biotechnological applications. We also investigate the molecular processes governing organisms’ interactions in their natural niche. Our interest is drawn towards the elucidation of the biogenesis pathways and cell-to-cee communication mechanisms driven by extracellular membrane vesicles (EMVs). EMVs are nanoparticles involved in a broad range of biological processes including horizontal DNA transfer, decoy for phages and antibiotics, disposal of waste material and surface remodeling, nutrient scavenging, bacterial killing, host response immunomodulation and delivery of bioactive compounds and thus are essential for inter- intra-species and inter-kingdom communication. Recently, interactomic-driven research has led us to the identification of highly conserved proteins sustaining EMV formation across species. In our current projects we propose to exploit the full potential of EMVs for the benefit of bacteria-plant interactions. To this end, we employ interactomics techniques consisting of affinity purification coupled with mass spectrometry, SPOT-membrane arrays, immunofluorescence and immunogold labelling microscopy and others to identify further protein-protein interactions underlying the biogenesis of EMVs in different bacterial species present in the plant holobiont. We induce the expression of these proteins found in the interactomic studies to modulate vesicle formation rate, size and amount. We intend to tailor the protein and metabolic cargo of EMVs by encapsulating nodulation factors, plant-priming molecules, nitrogen nitrogen-fixing enzymes or phytopathogen inhibitors. Thereby, EMVs can be used as organism-free biopesticides for phytopathogen killing or as bioinoculants to enhance nodulation, nitrogen fixation and ultimately plant growth. Nonetheless, EMV research go beyond the applications related to sustainable agriculture since novel molecular tools arising from this work can be applied in multiple disciplines, ranging from biomedicine (development of vaccines) to bioremediation (design of EMV-based xenobiotic degraders).
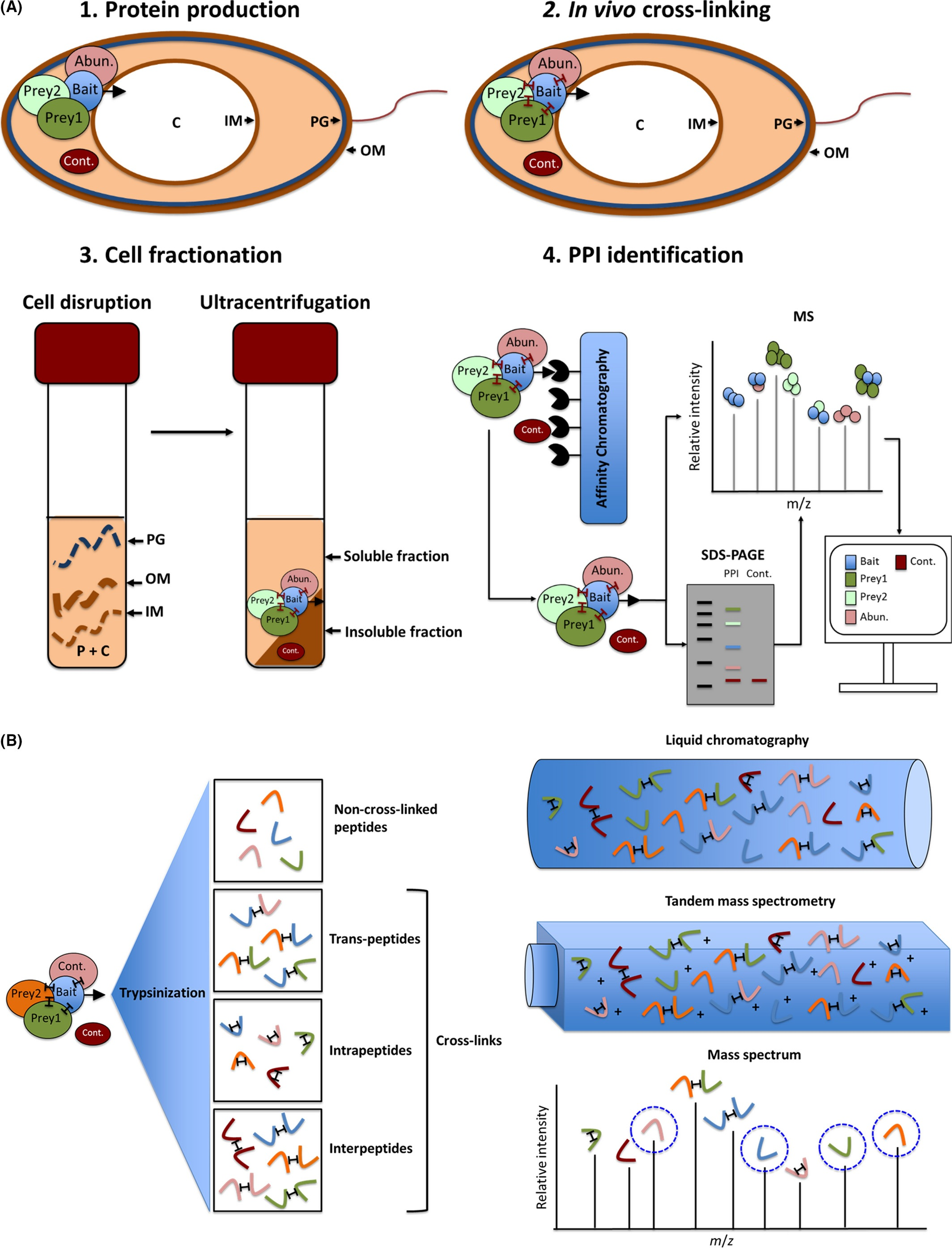
Figure 1. General interactomic workflow. The illustration depicts step by step the protein–protein interaction elucidation pathway via affinity chromatography copurification coupled with mass spectrometry (Borrero de Acuña, J. M. et al 2017).
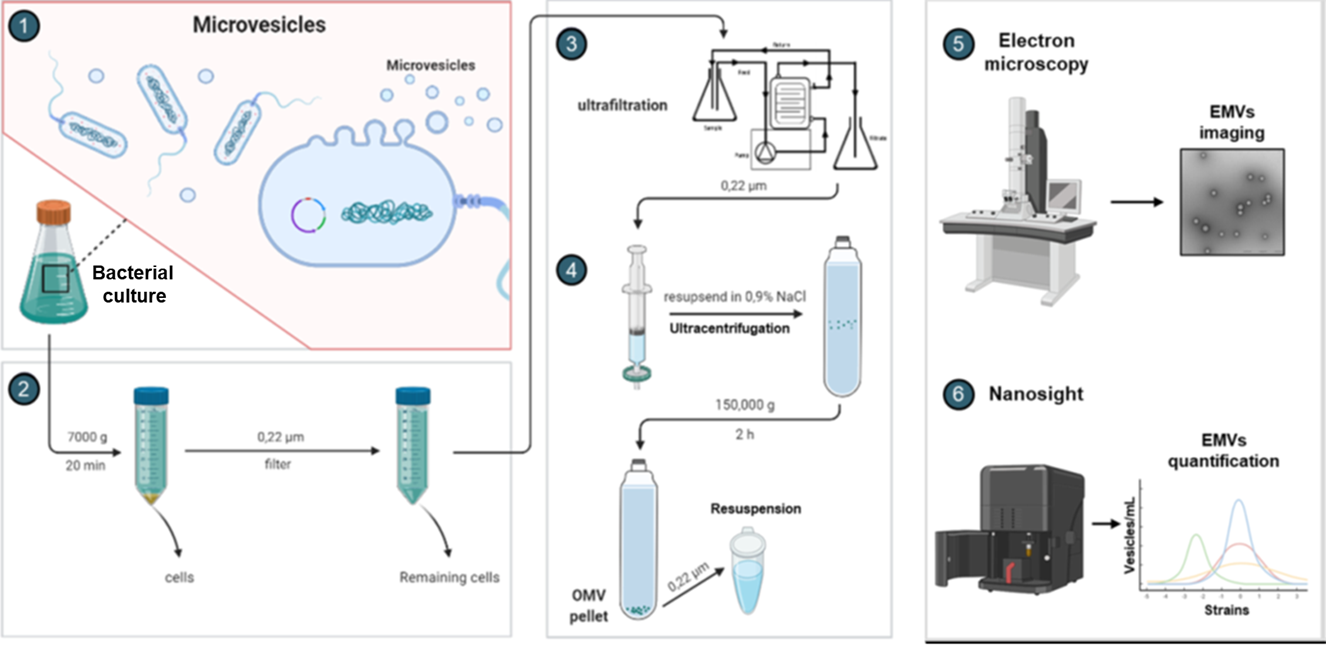
Figure 2. Experimental workflow designed for the isolation and characterisation of extracellular membrane vesicles (EMVs). (1) Growth of bacterial culture under standard conditions to produce EMVs. (2) Harvesting of cells and filtering of supernatants to remove remaining cells. (3) Ultrafiltration to remove impurities and concentrate EMV fractions. (4) Ultracentrifugation to collect EMV fractions. (5) Evaluation of the EMVs structure and integrity by electron microscopy. (6) Quantification of EMVs using scattering-light reliant Nanosight technology. Abbreviations: EMVs = extracellular membrane vesicles.
Research projects
- Title: Engineering extracelular membrane vesicles from rhizospheric bacteria for the development of biopesticides and plant-growth promoting agents.
- Funding Entity: Proyectos de Excelencia 2021. Secretaría General de Universidades, Investigación y Tecnología, Junta de Andalucía.
- Reference: ProyExcel_00450 Affiliation: Universidad de Sevilla Duration: diciembre 2022-noviembre 2025
- Funding Granted: 140.937,14€ Principal Investigator: Dr Jose Manuel Borrero de Acuña & Dra Patricia Bernal
- Title: Development of biopesticides based on membrane vesicles as a sustainable alternative to polluting chemical pesticides.
- Funding Entity: Ministerio de Ciencia e Innovación, Proyectos Estratégicos Orientados a la Transición Ecológica y a la Transición Digita 2021, Gobierno de España
- Reference: TED2021-130357B-I00 Affiliation: Universidad de Sevilla Duration: diciembre 2022-noviembre 2024
- Funding Granted: 253.000,00€ Principal Investigator : Dr Jose Manuel Borrero de Acuña & Dra Patricia Bernal
- Title: Enhancing the symbiotic cross-talk through rhizobial membrane vesicles.
- Funding Entity: Ministerio de Ciencia e Innovación, Proyectos de Generación de Conocimiento 2021, Gobierno de España
- Reference: PID2021-122395OA-I00 Affiliation: Universidad de Sevilla Duration: septiembre 2022-agosto 2026
- Funding Granted: 145.200,00€ Principal Investigator: Dr Jose Manuel Borrero de Acuña
- Title: Engineering membrane vesicles for fine-tuned modulation of rhizobia species interactions for enhanced nodulation and plant growth.
- Funding Entity: Junta de Andalucía, Consejería de transformación económica, industria, conocimiento y universidades
- Reference: EMERGIA20_00048 Affiliation: Universidad de Sevilla
- Duration: noviembre 2021- agosto 2025 Principal Investigator: Dr Jose Manuel Borrero de Acuña
- Title: Developing a rodent disease model for Pseudomonas aeruginosa infection in bronchiectasis for drug research.
- Funding Entity: Federal Ministry of Education and Research (Germany)
- Reference: 281361126/GRK2223 Affiliation: Technical University Braunschweig
- Duration: diciembre 2020- noviembre 2023 Principal Investigator: Dr Jose Manuel Borrero de Acuña
Pat
Selected publications
- Jiménez-Guerrero, I., López-Baena, F. J., Borrero-de Acuña, J. M.* & Pérez-Montaño, F. (2023). Membrane vesicle engineering with «à la carte» bacterial-immunogenic molecules for
organism-free plant vaccination. Microbial Biotechnology, (accepted; ahead of print). *Corresponding author - Borrero‐de Acuña, J. M., & Poblete‐Castro, I.* (2023). Rational engineering of natural polyhydroxyalkanoates producing microorganisms for improved synthesis and
recovery. Microbial Biotechnology, 16(2), 262-285. - Michel, A. M., Borrero‐de Acuña, J. M.*, Molinari, G., … & Jahn, D. (15 authors); (2022).Cellular adaptation of Clostridioides difficile to high salinity encompasses a compatible solute‐
responsive change in cell morphology. Environmental Microbiology, 24(3), 1499-1517.*Corresponding author - Borrero de Acuña, J. M., & Bernal, P.*; (2021). Plant holobiont interactions mediated by the type VI secretion system and the membrane vesicles: promising tools for a greener
agriculture. Environmental Microbiology, 23(4), 1830-1836. - Borrero‐de Acuña, J. M., Gutierrez‐Urrutia, I., Hidalgo‐Dumont, C., … & Poblete‐Castro, I.*; (14 authors); (2020). Channelling carbon flux through the meta‐cleavage route for improved
poly (3‐hydroxyalkanoate) production from benzoate and lignin‐based aromatics in Pseudomonas putida H. Microbial Biotechnology 14(6), 2385-2402. - Borrero-de Acuña, J. M., & Poblete-Castro, I.*; (2020). Expanding the Reach of Recombineering to Environmental Bacteria. Trends in Biotechnology, 38(7):684-685.
- Poblete-Castro, I., Aravena-Carrasco, C., Orellana-Saez, M., Pacheco, N., Cabrera, A., & Borrero-de Acuña, J. M.*; (2020). Engineering the osmotic state of Pseudomonas putida
KT2440 for efficient cell disruption and downstream processing of poly (3-hydroxyalkanoates). Frontiers in Bioengineering and Biotechnology, 8: 161. *Corresponding author - Borrero-de Acuña, J.M.*, Timmis, K.N., Jahn, M. & Jahn, D. (2017). Protein complex formation during denitrification by Pseudomonas aeruginosa. Microbial Biotechnology, 10(6),
1523-1534. *Corresponding author - Borrero-de Acuña, J.M.*, Hidalgo-Dumont, C., Pacheco, N., Cabrera, A. & Poblete-Castro, I.* (2017). A novel programmable lysozyme-based lysis system in Pseudomonas
putida for biopolymer production. Scientific Reports, 29;7(1): 4373. *Co-Corresponding authors - Borrero-de Acuña, J.M., Rohde, M., Wissing, J., … & Jahn, D.*; (9 authors). (2016) Protein network of the Pseudomonas aeruginosa denitrification apparatus. Journal of
Bacteriology. 198(9): 1401-13
PhD students
- Irene Herrero Gómez: Enhancing the symbiotic cross-talk through rhizobial membrane vesicles.
- Natalia Moreno de Castro: Engineering membrane vesicles for fine-tuned modulation of rhizobia species interactions for enhanced nodulation and plant growth.
- Carlos Azogue Palma: Engineering membrane vesicles for efficient production of industrially valuable chemicals.
riiaBernal
El sistema de secrecion de tipo VI (T6SS) de Pseudomonas putida. Potente arma de biocontrol para la protección de cultivos de interés
IP: Patricia Bernal
La línea de investigacion de la Dra. Bernal se centra en el estudio de los Sistemas de Secrecion de Tipo VI (T6SS) en Pseudomonas putida. P. putida es una bacteria del suelo con la capacidad de colonizar la raíz de diferentes plantas de cultivo proporcionando ventajas de crecimiento y al mismo tiempo protección contra patógenos; por ello esta cepa es considerada un excelente agente de control biológico (biocontrol). El biocontrol de las enfermedades producidas por patógenos de plantas se considera una excelente alternativa a los pesticidas químicos para proteger nuestros cultivos, ya que estos pueden provocar la contaminacion del subsuelo y la pérdida de la microbiota natural tanto del suelo como de la planta. Por esta razón, desde los organismos de financiacion se han priorizado diferentes iniciativas destinadas a promover una agricultura más sostenible, entre las que se encuadra el biocontrol. Para progresar en este campo de investigación, es clave entender de una manera global y completa los mecanismos moleculares de control biológico llevados a cabo por agentes de biocontrol conocidos y bien establecidos como Pseudomonas putida.
En P. putida, el T6SS ha sido establecido recientemente como un importante mecanismo de biocontrol que confiere a esta estirpe casi la totalidad de su capacidad biocontroladora (Bernal et al. 2017). El T6SS se considera una potente arma antibacteriana y P. putida lo usa eficientemente para aniquilar patógenos de plantas extremadamente deletéreos como Pseudomonas syringae o Agrobacterium tumefaciens. P. putida contiene tres T6SSs denominados K1-, K2- y K3-T6SS. El K1-T6SS se induce en fase de crecimiento estacionaria, secreta toxinas y destruye a una amplia gama de bacterias fitopatógenas tales como P. syringae, Xanthomonas campestris o Agrobacterium tumefaciens (Bernal et al. 2017, Bernal et al. 2021). Esta capacidad antimicrobiana ante fitopatógenos se ha observado tanto en experimentos in vitro como en experimentos in planta (Bernal et al. 2017, Bernal et al. 2021).
A nivel estructural, el T6SS es una nanomáquina contráctil formada por un componente de membrana, una cola (tubo y vaina contráctil) y una placa base. El componente de membrana ancla el sistema a la envoltura celular que reúne a los componentes de la placa base desde donde polimeriza la cola. Una vez el sistema está ensamblado, la vaina se contrae y el tubo con las proteínas efectoras se eyecta al interior de las células dianas (Fig. 3). Un componente elemental de este sistema, la proteína TssA, es indispensable para el ensamblaje de la vaina. A pesar de su función clave, las proteínas TssA exhiben una diversidad inesperada y existen en dos formas principales, una forma corta (TssAS) y una forma larga (TssAL). Mientras las proteínas TssAL interactúan con la proteína TagA para anclar el extremo distal de la vaina extendida, el mecanismo de estabilización de los T6SS que contienen TssAS siguía siendo desconocido. En un estudio reciente (Bernal et al., 2021) hemos identificado una nueva categoría de componentes estructurales (proteínas TagB y TagJ) que interactúan con proteínas TssAs y contribuyen al ensamblaje del T6SS estabilizando la vaina polimerizada desde la placa base. Además, hemos demostrado que la presencia de estos componentes es importante para la extensión completa de la vaina y para mantener una capacidad de disparo óptima. Asimismo, se ha visto que la asociación de las distintas formas de proteínas TssA con una clase diferente de proteínas estabilizadoras de la vaina resulta en T6SS que bien residen en la célula durante algún tiempo (TssAL-TagA) o disparan inmediatamente después de la extensión de la vaina (TssAS-TagB). Se propone, que esta diversidad en la dinámica de tiro podría contribuir a la especialización del T6SS para adaptarse a diferentes estilos de vida bacterianos en diversos nichos ambientales (Bernal et al., 2021).
Las líneas de investigación que se llevan a acabo en el laboratorio de la Dra. Bernal están dirigidas a conocer más fondo la función y el mecanismo de acción de estos sistemas antimicrobianos, lo quepermitir adaptarlos para desarrollar agentes de biocontrol altamente eficaces que sean más específicos y con una mayor potencia para erradicar patógenos de plantas y ser una herramienta para futuras aplicaciones biotecnológicas aplicadas a la agricultura.
En este video se resume el proyecto financiado por la Fundacion BBVA entre 2021-2022 y titulado: El superagente de biocontrol Pseudomonas putida KT2440 como medida sostenible de lucha frente a Xylella fastidiosa, la gran amenaza de nuestro olivar
Proyectos activos
- Título del Proyecto: Engineering extracelular membrane vesicles from rhizospheric bacteria for the development of biopesticides and plant-growth promoting agents.
- Entidad Financiadora: Proyectos de Excelencia 2021. Secretaría General de Universidades, Investigación y Tecnología, Junta de Andalucía.
- Referencia: ProyExcel_00450 Entidad Líder: Universidad de Sevilla Periodo: diciembre 2022-noviembre 2025
- Cuantía de la subvención: 140.937,14€ Investigadora Responsable: Dra Patricia Bernal & Jose Manuel Borrero de Acuña
- Título del Proyecto: Desarrollo de pesticidas biológicos basados en vesículas de membrana como alternativa sostenible a los pesticidas químicos altamente contaminantes (BIOPESTOMV).
- Entidad Financiadora: Ministerio de Ciencia e Innovación, Proyectos Estratégicos Orientados a la Transición Ecológica y a la Transición Digita 2021, Gobierno de España
- Referencia: TED2021-130357B-I00 Entidad Líder: Universidad de Sevilla Periodo: diciembre 2022-noviembre 2024
- Cuantía de la subvención: 253.000,00€ Investigadora Responsable: Dra Patricia Bernal & Jose Manuel Borrero de Acuña
- Título del Proyecto: Optimizando Pseudomonas putida como agente protector de cultivos: señales ambientales, especificidad y eficiencia del sistema de secreción tipo vi como arma de biocontrol
- Entidad Financiadora: Ministerio de Ciencia e Innovación, Proyectos de Generación de Conocimiento 2021, Gobierno de España
- Referencia: PID2021-123000OB-I00 Entidad Líder: Universidad de Sevilla Periodo: septiembre 2022-agosto 2025
- Cuantía de la subvención: 175.450,00€ Investigadora Responsable: Dra Patricia Bernal
- Título del Proyecto: Estudios de control biológico para la protección de cultivos a través del uso del sistema de secreción de tipo VI (T6SS) bacteriano
- Entidad Financiadora: Ministerio de Ciencia, Innovación y Universidades, Programa Ramón y Cajal Convocatoria 2019, Gobierno de España
- Referencia: RYC2019-026551-I Entidad Líder: Universidad de Sevilla
- Periodo: abril 2021-marzo 2026 Investigadora Responsable: Dra. Patricia Bernal
Publicaciones destacadas
-
Bernal, P*, Civantos, C, Pacheco-Sánchez, D, Quesada, JM, Filloux, A*, and Llamas*, MA (2023). Transcriptional organization and regulation of the Pseudomonas putida K1 Type VI secretion system gene cluster. Microbiology 169:001295. (*Co-autora de correspondencia).
- González-Magaña, A, Altuna,J, Queralt-Martín, M, Largo, E, Velázquez, C, Montánchez, I, Bernal, P, Alcaraz, A, Albesa-Jové, D (2022). The P. aeruginosa effector Tse5 forms ion-selective membrane pores that disrupt the membrane potential of intoxicated bacteria. Communications Biology. 5:1189.
- Bernal, P*, Eberl, L., de Jonge, R., Lepek, V.C. & Malone, J.G. (2021) Understanding plant-microorganism interactions to envision a future of sustainable agriculture. Editorial. Environ Microbiol. 23:1809-1811 (*Autora de correspondencia).
- Borrero de Acuña, JM, & Bernal, P* (2021). Plant holobiont interactions mediated by the type VI secretion system and the membrane vesicles: promising tools for a greener agriculture. Environ Microbiol. 23:1830-1836 (*Autora de correspondencia).
- Durán, D, Bernal, P, David Vazquez-Arias, Blanco-Romero, E, Garrido-Sanz, D, Redondo-Nieto, M, Rivilla, R and, Martin, M. (2021). Pseudomonas fluorescens F113 type VI Secretion Systems mediate bacterial killing and adaption to the rhizosphere microbiome. Scientific Report 11:5772-5785.
- Bernal, P, Furniss, CD, Fecht, S, Leung, RCY, Spiga, L, Mavridou, DAI & Filloux. (2021). A novel stabilization mechanism for the type VI secretion system sheath. Proc Natl Acad Sci. 118:e2008500118
- Bernal, P, Murillo-Torres, M & Allsopp, LP (2020). Integrating signals to drive T6SS killing. Environ Microbiol 22:4520-4523.
- Allsopp, LP, Bernal, P, Nolan, L & Filloux A. (2020). Causalities of War: The connection between T6SS & microbiota. Cell Microbiol 22:e13153.
- Bernal, P. *, Llamas, MA, Filloux, A. (2018). Type VI secretion Systems in plant-associated bacteria. Environ Microbiol 20:1-15 (*Autora de correspondencia).
- Bernal, P. *, Allsopp, L.P., Filloux, A., and Llamas M.A. (2017). The Pseudomonas putida T6SS is a plant warden against phytopathogens. ISME Journal 11:972-987 (*Autora de correspondencia).
quito
Papel del sistema de secreción de tipo VI y de las vesículas externas de membrana en la interacción rizobio-leguminosa.
Nuestro grupo de investigación tiene como objetivo profundizar en los mecanismos que determinan la especificidad y la eficiencia de la nodulación mediados por el sistema de secreción de tipo VI en los rizobios, usando como organismo modelo Sinorhizobium fredii USDA257, estirpe con uno de los mayores rangos de hospedador conocido. Además, en este proyecto se pretende determinar el papel de este sistema de secreción de proteínas en la competición con otras bacterias rizosféricas incluyendo algunas fitopatógenas. Los resultados esperados de este proyecto mejorarán el conocimiento existente sobre los mecanismos responsables de la compatibilidad rizobio-leguminosa, así como el posible uso de los rizobios no solo como agentes biofertilizantes sino también como agentes de biocontrol.
Además, en colaboración con el Dr. Borrero de Acuña, nuestro grupo propone aprovechar todo el potencial de las vesículas externas de membrana en beneficio de las interacciones rizobio-leguminosa mediante una aproximación biotecnológica que evita el uso de OMG. Para ello, además de determinar el cargo proteínico y metabólico de las vesículas externas de membrana en diferentes rizobacterias (S. fredii HH103 y Rhizobium tropici CIAT 899) y estadios simbióticos, se pretende adaptar en parte la carga proteica y metabólica de estas estructuras membranosas para que encapsulen mayoritariamente moléculas de interés biotecnológico, como factores de nodulación, fitohormonas, etc. De esta forma, el objetivo final es mejorar la fijación de nitrógeno y el crecimiento de las plantas mediante el uso de vesículas cargadas a la carta, sin la necesidad de emplear la totalidad del microorganismo.
Proyectos activos
Título del Proyecto: ¿Antibiosis o simbiosis? Caracterizando el sistema de secreción de tipo VI de Sinorhizobium fredii USDA257.
Entidad Financiadora: Plan Estatal 2017-2020 Retos – Proyectos I+D+i. Ministerio de Ciencia e Innovación
Referencia: PID2020-118279RA-I00 Entidad Líder: Universidad de Sevilla Periodo: septiembre 2021-agosto 2024
Cuantía de la subvención: 157.200 € Investigadora Responsable: Francisco Pérez Montaño
Título del Proyecto: Legumes in biodiversity-based farming systems in Mediterranean basin.
Entidad Financiadora: Partnership for Research and Innovation in the Mediterranean Area (PRIMA)
Referencia: 2019-SECTION2-19 Entidad Líder: Centro IFAPA Periodo: diciembre 2020-noviembre 2023
Cuantía de la subvención: 150.000 € Investigadora Responsable: Dulce Nombre Rodríguez Navarro
Publicaciones últimos 5 años (*Autoría preferente)
Jiménez-Guerrero I, López-Baena, FJ, Borrero-de Acuña, JM, Pérez-Montaño F*. (2023). Membrane vesicle engineering with «à la carte» bacterial-immunogenic molecules for organism-free plant vaccination. Microbial Biotechnology. 10.1111/1751-7915.14323
Fuentes-Romero F, Moyano-Bravo I, Ayala-García P, Rodríguez-Carvajal MA, Pérez-Montaño F, Acosta-Jurado S, Ollero FJ, Vinardell JM. (2023). Non-Ionic Osmotic Stress Induces the Biosynthesis of Nodulation Factors and Affects Other Symbiotic Traits in Sinorhizobium fredii HH103. 10.3390/biology12020148.
Ayala-García P, Jiménez-Guerrero I, Jacott CN, López-Baena FJ, Ollero FJ, Del Cerro P, Pérez-Montaño F*. (2022). The Rhizobium tropici CIAT 899 NodD2 protein promotes symbiosis and extends rhizobial nodulation range by constitutive nodulation factor synthesis. 10.1093/jxb/erac325.
de la Osa C, Pérez‐López J, Feria AB, Baena G, Marino D, Coleto I, Pérez-Montaño F, Gandullo J, Echeverria C, García-Nauriño S, Monreal, J. A. (2022). Knock‐down of phosphoenolpyruvate carboxylase 3 negatively impacts growth, productivity and responses to salt stress in sorghum (Sorghum bicolor L.). 10.1111/tpj.15789.
Fuentes-Romero F, Navarro-Gómez P, Ayala-García P, Moyano-Bravo I, López-Baena FJ, Pérez-Montaño F, Ollero FJ, Acosta-Jurado A, Vinardell, J. M. (2022). The nodD1 Gene of Sinorhizobium fredii HH103 Restores Nodulation Capacity on Bean in a Rhizobium tropici CIAT 899 nodD1/nodD2 Mutant, but the Secondary Symbiotic Regulators nolR, nodD2 or syrM Prevent HH103 to Nodulate with This Legume. Microorganisms. 10.3390/microorganisms10010139.
Jiménez-Guerrero I, Moreno-de Castro N, Pérez-Montaño F*. (2021). One door closes, another opens: when nodulation impairment with natural hosts extends rhizobial host-range. Environmental Microbiology. 10.1111/1462-2920.15353.
Aharon E; Mookherjee A; Pérez-Montaño F; Mateus da Silva G; Sathyamoorthy R; Burdman, S; Jurkevitch E. (2021). Secretion systems play a critical role in resistance to predation by Bdellovibrio bacteriovorus. Research in Microbiology. 10.1016/j.resmic.2021.103878.
del Cerro P, Ayala-García P, Buzón P, Castells-Graells R, López-Baena FJ, Ollero FJ, Pérez-Montaño F*. (2019). OnfD, an AraC-type transcriptional regulator of Rhizobium tropici CIAT 899 involved in Nod factor synthesis and symbiosis. Applied and Environmental Microbiology. 10.1128/aem.01297-20.
Jiménez-Guerrero I, Acosta-Jurado S, Medina C, Ollero FJ, Alias-Villegas C, Vinardell JM, Pérez-Montaño F, López-Baena FJ. (2020). The Sinorhizobium fredii HH103 Type III secretion system effector NopC blocks nodulation with Lotus japonicus Gifu. Journal of Experimental Botany. 10.1093/jxb/eraa297.
Martinelli F, Ollero FJ, Giovino A, Perrone A, Bekki A, Sikora S, Nabbout RE, Bouhadida M, Yucel D, Bazzicalupo M, Mengoni A, Pérez-Montaño F*. (2020). Proposed research for innovative solutions for chickpeas and beans in a climate change scenario: the Mediterranean Basin. Sustainability. 10.3390/su12041315.
Acosta-Jurado S, Alías-Villegas C, Almozara A, Espuny MR, Vinardell JM, Pérez-Montaño F*. (2020). Deciphering the symbiotic significance of quorum sensing systems of Sinorhizobium fredii HH103. Microorganisms. 10.3390/microorganisms8010068.
Jiménez-Guerrero I, Pérez-Montaño F*, da Silva GM, Wagner N, Shkedy D, Zhao M, Pizarro L, Bar M, Walcott R, Sessa G, Pupko T, Burdman S. (2019). Show me your secret(ed) weapons: a multifaceted approach reveals a wide arsenal of type III-secreted effectors in the cucurbit pathogenic bacterium Acidovorax citrulli and novel effectors in the Acidovorax genus. Molecular Plant Pathology. 10.1111/mpp.12877.
Yang R, Santos-Garcia D, Pérez-Montaño F, Zhao M, Jimenez-Guerrero, I, Da Silva GM, Rosenberg T, Chen G, Plaschkes I, Morin S, Walcott R, Burdman S. (2019). Complete assembly of the genome of an Acidovorax citrulli strain reveals a naturally occurring plasmid in this species. Frontiers in Microbiology. 10.3389/fmicb.2019.01400.
del Cerro P, Ayala-García P, Jiménez-Guerrero I, López-Baena FJ, Vinardell JM, Megías M, Hungria M, Gil-Serrano A, Pérez-Montaño F, Ollero FJ. (2019). The non-flavonoid inducible nodA3 and the flavonoid regulated nodA1 genes of Rhizobium tropici CIAT 899 guarantee nod factor production and nodulation of different host legumes. Plant and Soil. 10.1007/s11104-019-04073-2.
del Cerro P, Megias M, López-Baena FJ, Gil-Serrano A, Perez-Montano F, Ollero FJ. (2019). Osmotic stress activates nif and fix genes and induces the Rhizobium tropici CIAT 899 Nod factor production via NodD2 by up-regulation of the nodA2 operon and the nodA3 gene. PLoS ONE. 10.1371/journal.pone.0213298.
Doctorandos en formación
Irene Herrero Gómez: Potenciando el diálogo simbiótico a través de las vesículas de membrana externa rizobianas. Co-supervisado con el Dr. Borrero de Acuña.
Natalia Moreno de Castro: Ingeniería de vesículas de membrana externa rizobianas para la mejora de la nodulación y el crecimiento vegetal. Co-supervisado con el Dr. Borrero de Acuña.
Paula Ayala García: Estudio dual de los mecanismos moleculares y fisiológicos responsables de la nodulación entre Rhizobium tropici CIAT 899 y las leguminosas Phaseolus vulgaris y Lotus japonicus. Co-supervisado con el Dr. Ollero Márquez.
Doctorandos egresados
Pablo del Cerro Sánchez: «Caracterización de genes de Rhizobium tropici CIAT899 implicados en la biosíntesis de los factores de nodulación independiente de la activación por flavonoides e inducidos por estrés osmótico». 2019. Co-supervisado con el Dr. Ollero Márquez.
