
Foto de grupo.De izquierda a derecha. Francisco García del Portillo, M. Graciela Pucciarelli, Pablo García, Estel Ramos, Diana Barroso, Gadea Rico, Daniela Gargano y Noelia López.
Muchas enfermedades que impactan la salud del hombre y animales están causadas por bacterias que invaden y colonizan el nicho intracelular de células eucariotas. Entre estas enfermedades destacan, entre otras, tuberculosis, fiebre tifoidea, listeriosis, clamidiosis, brucelosis, rickettiosis, fiebre Q y legionelosis. Algunas bacterias patógenas que producen estas patologías, como las delos géneros Rickettsia spp., Chamydia spp. y Coxiella spp., son incapaces de crecer y proliferar fuera de la célula eucariota.
De enorme interés de investigación son también las bacterias patógenas intracelulares‘facultativas’ que colonizan distintos tipos celulares eucariotas además de ambientes diversos fuera del hospedador. Los datos obtenidos hasta la fecha muestran la existencia de sistemas de regulación que se encargan de reprogramar la fisiología de la bacteria durante la transición desde el ambiente extracelular al intracelular, o viceversa. Nuestro grupo aborda como principal objetivo el conocimiento de estas pautas de regulación y, por ende, los mecanismos de adaptación al estilo de vida intracelular una vez el patógeno ha invadido la célula eucariota. Para acometer esta empresa, utilizamos dos bacterias con distinta envoltura y variado modo de vida intracelular. Por un lado, la bacteria Gram-positiva Listeria monocytogenes, agente causante de la listeriosis. L. monocytogenes atraviesa barreras de defensa naturales como el epitelio intestinal, la barrera hematoencefálica y la placenta. Como segundo patógeno modelo, la bacteria Gram-negativa Samonella enterica serovar Typhimurium (S. Typhimurium), agente causante de gastroenteritis y, eventualmente, de enfermedad sistemática si existe riesgos adicionales como coinfección o alteraciones en defensas del hospedador. Ambos patógenos inician la infección en ganado y humanos tras el consumo de alimentos contaminados
La pared celular bacteriana y la adaptación al ambiente intracelular eucariota
Una estructura celular clave en el mantenimiento de la forma e integridad de casi todas las bacterias es la ‘pared’ o envuelta. Dentro de la pared es la macromolécula de peptidoglicano, también conocida como ‘mureína’, la que asegura la forma e integridad celular.Históricamente, la bioquímica del peptidoglicano ha sido estudiada intensamente por ser blanco de antibióticos como los del grupo de beta-lactámicos. Estudios más recientes indican que el peptidoglicano es una señal de alarma para sistema de defensa innatos del hospedador. Así, se han caracterizado receptores dispuestos en el citosol de células eucariotas, como Nod1 y Nod2, que reconocen ‘patrones moleculares’ presentes en fragmentos del peptidoglicano. Curiosamente, este reconocimiento se da en el interior de la célula eucariota, indicando que la evolución parece haber diseñado defensas para el control de infecciones bacterianas intracelulares. De interés, determinados patógenos bacterianos intracelulares han evolucionado realizando modificaciones en el peptidoglicano que influyen en su reconocimiento por enzimas de defensa (pe. lisozima) o receptores tipo Nod. En el contexto de nuestra investigación, estamos también interesados en una gran familia de proteínas unidas covalentemente al peptidoglicano en el género Listeria. La gran mayoría de estas proteínas de superficie (aparecen en una media de 40 en todas las especies y estirpes de este género con genoma secuenciado) son exclusivas de Listeria, existiendo un numero apreciable de las mismas que están presentes sólo en especies patógenas. La función de aproximadamente el 90% de estas proteínas es desconocida, aunque se postula podrían estar involucradas en la adaptación de Listeria a ambientes diversos.
Nuestra investigación en L. monocytogenes y S. enterica serovar Typhimurium
Los abordajes experimentales que utiliza nuestro grupo tienen un denominador común, el “aislamiento de bacteria de células eucariotas infectadas en cultivo”. Esta metodología es laboriosa en lo que respecta al número de células eucariotas que hay que infectar para obtener la cantidad de material bacteriano necesaria para realizar biología molecular. Como ejemplo ilustrativo, la obtención de 107 bacterias intracelulares (el equivalente a 10 microlitros de un cultivo bacteriano que ha crecido una noche en medio de laboratorio!!!) supone infectar aproximadamente del orden de 107 fibroblastos o células epiteliales. Alcanzar tal cantidad de células eucariotas en cultivo supone al menos una semana de trabajo. Cuando el experimento tiene por objeto la purificación de peptidoglicano sintetizado por la bacteria intracelular, estos números se incrementan en varios órdenes de magnitud. A pesar de estas dificultades, este abordaje experimental es el único que permite obtener información sobre la biología del patógeno durante su ciclo de infección intracelular. Así, hemos obtenido logros como el transcriptoma de S. Typhimurium cuando persiste en un estado intracelular de no crecimiento; la estructura del peptidoglicano que S. Typhimurium sintetiza en el interior de células epiteliales; el proteoma de la pared de L. monocytogenes aislada de células epiteliales infectadas; y, el perfil de expresión en tiempo real y a lo largo de distintas fases de la infección intracelular de 56 RNA pequeños reguladores (sRNA) de S. Typhimurium.
La información de‘carácter global’obtenida en esos estudios nos permite en la actualidad abordar con similar metodología preguntas que ahora dirigimos a proteínas, RNA reguladores y procesos concretos. Como ejemplo, hemos descifrado una regulación que actúa sobre una proteína de L. monocytogenes unida a peptidoglicano que el patógeno induce en el ambiente intracelular. El aumento de los niveles de esta proteína requiere la unión de un sRNA regulador a una región 5’-UTR (región no traducida del mRNA) de una variante del tránscrito del gen diana. En S. Typhimurium estamos estudiando enzimas implicadas en la síntesis, remodelación e hidrólisis del peptidoglicano en bacteria intracelular, siempre teniendo como referencia bacteria crecida en medios de laboratorio. Dentro de este grupo de enzimas hemos identificado algunas exclusivas del género Salmonella. Curiosamente, algunas de ellas son reguladas positivamente en el ambiente intracelular.
Señalar igualmente nuestro interés por conocer la ‘biología celular’de la infección intracelular, centrándonos en un modelo de infección persistente en el que la bacteria no prolifera en la célula eucariota. Los datos obtenidos hasta la fecha implican a la maquinaria autofágica de la célula hospedadora como regulador del crecimiento de S. Typhimurium. En el caso de la infección persistente, la autofagia del patógeno intracelular muestra características distintivas a lo descrito en la literatura en otros modelos de infección.
Destacar como mensaje final las extraordinarias diferencias que observamos en muchos procesos cuando estudiamos estas bacterias en el ambiente intracelular y extracelular. El entender cómo, cuándo, y quién es el responsable de estos cambios mantendrá sin duda nuestro entusiasmo por esa ‘vida’ todavía tan misteriosa que determinadas bacterias patógenas desarrollan en el interior de nuestras células.
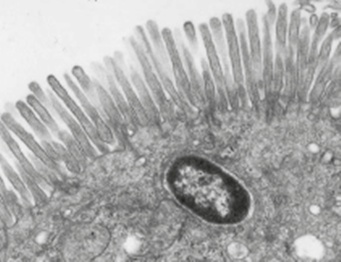
Figura1. Imagen de Salmonella enterica serovar Typhimurium tras invadir un enterocito del epitelio intestinal de ratón. Nótese que la bacteria se localiza en un compartimento vacuolar, también conocido como fagosoma ó SCV, por ‘Salmonella-containing vacuole’. A diferencia de este estilo de vida intracelular, Listeria monocytogenes utiliza proteínas que degradan la membrana fagosomal, proliferando posteriormente en el citosol de la célula infectada.
Bibliografía representativa
Ortega AD, Quereda JJ, Pucciarelli, MG y García-del Portillo, F. (2014). Non-coding RNA regulation in pathogenic bacteria located inside eukaryotic cells. Front CellInfectMicrobiol. (en prensa)
Quereda JJ, Ortega AD, Pucciarelli, MG y García-del Portillo, F. (2014). The Listeria small RNA Rli27 regulates a cell wall protein inside eukaryotic cells by targeting a long 5′-UTR variant. PLoSGenetics (en prensa)
Hernández , SB, Cava F, Pucciarelli MG, García-del Portillo F., de Pedro MA y Casadesús J. (2014). Bile-induced peptidoglycan remodelling in Salmonella enterica. Environ. Microbiol. doi: 10.1111/1462-2920.12491.
Bécavin C, Bouchier C, Lechat P, Archambaud C, Creno S, Gouin E, Wu Z, Kühbacher A, Brisse S, Pucciarelli MG, García-Del Portillo F., Hain T, Portnoy DA, Chakraborty T, Lecuit M, Pizarro-Cerdá J, Moszer I, Bierne H y Cossart P. (2014) Comparison of widely used Listeria monocytogenes strains EGD, 10403S and EGD-e highlights genomic divergence underlying differences in pathogenicity. mBIO doi: 10.1128/mBio.00969-14.
Mariscotti JF, Quereda JJ, García-Del Portillo F, Pucciarelli MG. (2014) The Listeria monocytogenes LPXTG surface protein Lm01413 is an invasin with capacity to bind mucin. Int. J. Med. Microbiol. 304: 393-404.
Quereda JJ y Pucciarelli MG (2014) Deletion of the membrane protein Lmo0412 increases the virulence of Listeria monocytogenes. MicrobesInfect. 16: 623-632.
Núñez-Hernández C, Alonso A, Pucciarelli MG, Casadesús J y García-Del Portillo F. (2014). Dormant intracellular Salmonella discriminates among SPI-2 effectors to persist inside fibroblasts. Infect. Immun. 82: 221-232.
Gonzalo-Asensio J, Ortega AD, Rico-Pérez G, Pucciarelli MG y García-Del Portillo F (2013). A Novel antisense RNA from the Salmonella virulence plasmid pSLT expressed by non-growing bacteria inside eukaryotic cells. PLoS One 8(10):e77939. doi: 10.1371/journal.pone.0077939.Microbiol. 16: 87-92.
Silva, IJ, Ortega AD, Viegas SC, García-del Portillo F (*) y Arraiano CM. (2013). An RpoS-dependent sRNA regulates the expression of a chaperone involved in protein folding. RNA 19: 1253-1265. (*) co-corresponding author.
Hernández, S.B., Ayala, J.A., Rico-Pérez, G., García-del Portillo, F., Casadesús, J. (2013) Increased bile resistance in Salmonella enterica mutants lacking Prc periplasmic protease Int. Microbiol. 2013 Jun;16(2):87-92.
Núñez-Hernández C, Tierrez A, Ortega AD, Pucciarelli MG, Godoy M, Esiman B, Casadesús J y García-del Portillo, F. (2013). Genome expression analysis of non-proliferating intracellular Salmonella unravels an acid pH-dependent PhoP-PhoQ response essential for dormancy. Infect. Immun.81:154-165.
Ortega, A.D., Gonzalo-Asensio, J. y García-del Portillo, F. (2013). Dynamics of Salmonella small RNA expression in non-growing bacteria located inside eukaryotic cells. RNA Biology 9: 469-488.
García-del Portillo F, Calvo E, D’Orazio V y Pucciarelli MG (2011). Association of ActA to peptidoglycan revealed by cell wall proteomics of intracelular Listeria monocytogenes. J. Biol. Chem. 286: 34675-34689.
Aiastui A, Pucciarelli MG y García-del Portillo F. (2010). Salmonella invades fibroblasts by multiple routes differing from the entry into epithelial cells. Infect. Immun. 78: 2700-2713.
