
Foto de grupo. De izquierda a derecha. Luis M. Mateos, Alfonso Gonzalo, Miriam Arienza, Almudena F. Villadangos, José A. Gil.
El grupo de investigación dirigido por los doctores José A. Gil y Luis M. Mateos, denominado “Biología Molecular de Corinebacterias” (http://microbio.unileon.es/wordpress), estudia diversos aspectos de la Biología Molecular de la corinebacteria Corynebacterium glutamicum, una actinobacteria que debe el epíteto de especie “glutamicum” a su capacidad para producir ácido glutámico. Durante los últimos quince años, el grupo ha centrado su investigación en: A) el estudio de los procesos de división celular en C. glutamicum, con objeto de usar esos conocimientos para el diseño de agentes antimicrobianos que controlen enfermedades producidas por corinebacterias y micobacterias patógenas; B) el análisis de la resistencia a arsénico en C. glutamicum con el objetivo de conocer los sistemas desintoxicantes de metales y su efecto en el estrés celular, y la consecución de microorganismos capaces de ser utilizados como biocontenedores de metales pesados y aplicados en biorremediación.
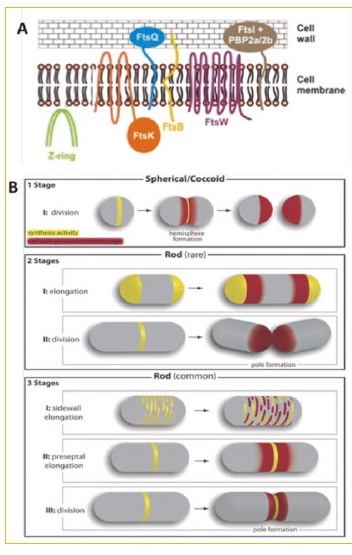 Figura1. A) Maquinaria de división minimalista de Corynebacterium glutamicum. B) Modelos de elongación/división en diferentes bacterias. (1 stage). Crecimiento a nivel del septo de división en bacterias cocoides que carecen de MreB como Streptococcus pneumoniae (2 stages). Crecimiento polar en bacterias bacilares que carecen de MreB como C. glutamicom o Mycobacterium tuberculosis. (3 stages) Crecimiento dirigido por MreB a lo largo de la pared lateral de B. subtilis o E. coli. El color amarillo muestra los sitios de síntesis de nuevo peptidoglucano y el color rojo muestra el sitio donde se inserta el nuevo peptidoglucano. (http://jcb.rupress.org/content/179/3/381.full)
Figura1. A) Maquinaria de división minimalista de Corynebacterium glutamicum. B) Modelos de elongación/división en diferentes bacterias. (1 stage). Crecimiento a nivel del septo de división en bacterias cocoides que carecen de MreB como Streptococcus pneumoniae (2 stages). Crecimiento polar en bacterias bacilares que carecen de MreB como C. glutamicom o Mycobacterium tuberculosis. (3 stages) Crecimiento dirigido por MreB a lo largo de la pared lateral de B. subtilis o E. coli. El color amarillo muestra los sitios de síntesis de nuevo peptidoglucano y el color rojo muestra el sitio donde se inserta el nuevo peptidoglucano. (http://jcb.rupress.org/content/179/3/381.full)División celular
Hemos estudiado el proceso de división/elongación celular de C. glutamicum ya que posee una maquinaria de división “minimalista” cuando se compara con la maquinaria de Escherichia coli (Fig. 1.A), es decir, carece de muchas de las proteínas (FtsA, FtsL, FtsN, AmiC, MinCDE, ZipA, MreB…) que se consideran esenciales en la maquinaria de división celular de E. coli. Durante estos últimos años hemos estudiado el papel de la proteína DivIVA en el proceso de elongación celular (Letek et al., 2008; Valbuena et al., 2007), así como el papel de las serín-treonin-proteín quinasas en dicho proceso.
(Fiuza et al., 2008a y 2008b). Asimismo describimos la presencia de un filamento intermedio (RsmP) en C. glutamicum regulado por fosforilación e implicado en el mantenimiento de la morfología bacilar (Fiuza et al., 2010) glutamicum muestra un modelo de crecimiento “raro” que los diferencia de los modelos clásicos de E. coli, Bacillus subtilis o Streptococcus pneumoniae (Fig. 1.B). Los resultados obtenidos con C. glutamicum nos permitirán diseñar compuestos que inhiban la división celular en organismos patógenos relacionados como C. diphtheriae o M. tuberculosis.
Resistencia bacteriana a arsénico
La otra línea de investigación se ha basado en la interacción arsénico-bacterias; el arsénico ha estado presente en la atmósfera desde la formación de la Tierra y los seres vivos han estado sometidos de forma continua a dicho agente, por lo que es bastante frecuente la presencia de sistemas de desintoxicación o resistencia a arsénico (Mateos et al., 2006). C. glutamicum no es una excepción y presenta genes de resistencia a arsénico que se agrupan en dos operones ars, codificando cada uno de ellos para un regulador/represor (ArsR), una proteína arseniato reductasa (ArsC) y una arsenito permeasa (Acr3).
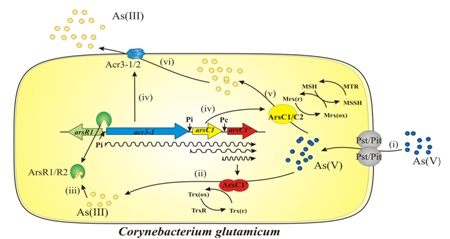
Figura 2. Modelo de resistencia a arsénico en C. glutanicum. (i) Entrada de arseniato [As(V)]; (ii) expresión constitutiva de la proteína ArsC1 y reducción de As(V) a arsenito [As(III)]; (iii) unión de As(III) a los reguladores (ArsR1/R2) y desrepresión del operón ars; (iv) transcripción de los genes que codifican para las arseniato reductasas (ArsC1/C2/C1′); (v) reducción de As(V) por reductasas ArsC1 y ArsC2 usando el sistema MSH/Mrx1 y (vi) liberación de As(III) formado en la célula a través de las arsenito permeasas Acr3-1/2. (Viladangos et al., 2011).
A partir de los análisis de los genes y las correspondientes proteínas desintoxicantes, se obtuvieron clones mutantes y recombinantes de C. glutamicum capaces de acumular hasta 30 veces más arsénico que la cepa silvestre y que podrían ser utilizados en procesos de biorremediación (Ordóñez et al., 2012; Villadangos et al., 2010). Igualmente se han utilizado algunas cepas mutantes de C. glutamicum para conseguir bioacumuladores específicos de alguna de las formas inorgánicas de arsénico, bien arsenito o arseniato (Villadangos et al., 2014).En ausencia de arsenito (la forma reducida del arsénico inorgánico), el regulador ArsR está reprimiendo la expresión del resto de los genes; en su presencia, ArsR tiene mucha afinidad por arsenito y se libera del operador, permitiendo de esta forma la transcripción de los dos genes restantes (Villadangos et al., 2011). Las enzimas arseniato reductasas intracelulares reducen el arseniato a arsenito (este último es muy tóxico), el cual es rápidamente liberado al exterior de la célula por las arsenito permeasas transmembranales (Fu et al., 2009; Villadangos et al., 2012) (Fig. 2).
Actualmente, el objetivo principal de la investigación está centrado en el estudio de los mecanismos diferenciales de defensa antioxidante que presentan las actinobacterias, utilizando como modelos C. glutamicum y Rhodococcus equi (modelo intermedio entre C. glutamicum y M. tuberculosis). El sistema Redox más ubicuo en las células es el constituido por el sistema tiorredoxina (Trx)/tiorredoxina reductasa (TrxR), presente en procariotas y eucariotas y constituyendo una primera línea de defensa frente a la oxidación. Sin embargo, los procesos evolutivos han permitido la aparición de otros sistemas Redox alternativos que son de diferente naturaleza dependiendo del organismo de que se trate. Estos sistemas alternativos dependen de redoxinas específicas y de compuestos de bajo peso molecular. Nuestro grupo de investigación ha sido pionero en la descripción de un nuevo sistema Redox celular exclusivo de actinobacterias que incluye el pseudoazúcar de bajo peso molecular micotiol (MSH) y la redoxina correspondiente capaz de reconocer de forma específica MSH; esta redoxina se denominó micorredoxina (Mrx) (Ordóñez et al., 2009). La “aparición” de este buffer Redox diferencial hace que el par MSH/Mrx y/o cualquiera de las proteínas que interactúan con MSH/Mrx (micotiolación) puedan constituir nuevas dianas antimicrobianas susceptibles de ser utilizadas de manera específica para este grupo de microorganismos (Van Laer et al., 2012; Van Laer et al., 2013). Los estudios basados en el grado de importancia y/o esencialidad de cada una de estas dianas constituyen en estos momentos el grueso de nuestra línea de investigación.
Durante los últimos años nuestro grupo está colaborando con otros grupos de investigación que tienen intereses científicos comunes, y liderados por los investigadores: William Margolin (USA), Klas Flardh (Suecia), Virginie Molle (Francia), Richard Daniel (UK), Joris Messens (Bélgica), Joern Kalinowsky (Alemania), Barry P. Rosen (USA), Juan Ayala (Madrid) y Jose A. Ainsa (España).
Fiuza M, Canova MJ, Patin D, Letek M, Zanella-Cleon I, Becchi M, Mateos LM, Mengin-Lecreulx D, Molle V, Gil JA. (2008a). The MurC ligase essential for peptidoglycan biosynthesis is regulated by the serine/threonine protein kinase PknA in Corynebacterium glutamicum. J Biol Chem. 283:36553-36563.
Fiuza M, Canova MJ, Zanella-Cleon I, Becchi M, Cozzone AJ, Mateos LM, Kremer L, Gil JA, Molle V. (2008b). From the characterization of the four serine/threonine protein kinases (PknA/B/G/L) of Corynebacterium glutamicum toward the role of PknA and PknB in cell division. J Biol Chem. 283:18099-18112.
Fiuza M, Letek M, Leiba J, Villadangos AF, Vaquera J, Zanella-Cleón I, Mateos LM, Molle V, Gil JA. (2010). Phosphorylation of a novel cytoskeletal protein (RsmP) regulates rod-shape morphology in Corynebacterium glutamicum. J Biol Chem. 285:24387-24397.
Fu HL, Meng Y, Ordóñez E, Villadangos AF, Bhattacharjee H, Gil JA, Mateos LM, Rosen BP. (2009). Properties of arsenite efflux permeases (Acr3) from Alkaliphilus metalliredigens and Corynebacterium glutamicum. J Biol Chem. 284:19887-19895.
Letek M, Ordóñez E, Vaquera J, Margolin W, Flärdh K, Mateos LM, Gil JA. (2008). DivIVA is required for polar growth in the MreB-lacking rod-shaped actinomycete Corynebacterium glutamicum. J Bacteriol. 190:3283-3292.
Letek M, Fiuza M, Villadangos AF, Mateos LM, Gil JA. (2012). Cytoskeletal proteins of actinobacteria. International. J Cell Biol. doi:10.1155/2012/905832.
Mateos LM, Ordóñez E, Letek M, Gil JA. (2006). Corynebacterium glutamicum as a model bacterium for the bioremediation of arsenic. Int Microbiol. 9:207-215.
Mateos LM, Letek M, Villadangos AF, Fiuza M, Ordóñez E, Gil JA. (2011). Chapter 6: Corynebacterium. In: Molecular detection of human bacterial pathogens. Edited by: Dongyou Liu. Taylor y Francis CRC Press. USA. ISBN: 978-1439812389.
Ordóñez E, Van Belle K, Roos G, De Galan S, Letek M, Gil JA, Wyns L, Mateos LM, Messens J. (2009). Arsenate reductase, mycothiol, and mycoredoxin concert thiol/disulfide exchange. J Biol Chem. 284:15107-15116.
Ordóñez E, Villadangos AF, Fiuza M, Pereira FJ, Gil JA, Mateos LM, Aller AJ. (2012). Modelling of arsenate retention from aqueous solutions by living coryneform double-mutant bacteria. Environm Chem. 9:121-129.
Valbuena N, Letek M, Ordóñez E, Ayala J, Daniel RA, Gil JA, Mateos LM. (2007). Characterization of HMW-PBPs from the rod-shaped actinomycete Corynebacterium glutamicum: peptidoglycan synthesis in cells lacking actin-like cytoskeletal structures. Mol Microbiol. 66:643-657
Van Laer K, Buts L, Foloppe N, Vertommen D, Van Belle K, Wahni K, Roos G, Nilsson L, Mateos LM, Rawat M, van Nuland NA, Messens J. (2012). Mycoredoxin-1 is one of the missing links in the oxidative stress defence mechanism of Mycobacteria. Mol Microbiol. 86:787-804.
Van Laer K, Dziewulska AM, Fislage M, Wahni K, Hbeddou A, Collet JF, Versées W, Mateos LM, Tamu Dufe V, Messens J. (2013). NrdH-redoxin of Mycobacterium tuberculosis and Corynebacterium glutamicum dimerizes at high protein concentration and exclusively receives electrons from thioredoxin reductase. J Biol Chem. 15:7942-7955.
Villadangos AF, Ordóñez E, Muñoz MI, Pastrana IM, Fiuza M, Gil JA, Mateos LM, Aller AJ. (2010). Retention of arsenate using genetically modified coryneform bacteria and determination of arsenic in solid samples by ICP-MS. Talanta. 80:1421-1427.
Villadangos AF, Van Belle K, Wahni K, Dufe VT, Freitas S, Nur H, De Galan S, Gil JA, Collet JF, Mateos LM, Messens J. (2011). Corynebacterium glutamicum survives arsenic stress with arsenate reductases coupled to two distinct redox mechanisms. Mol Microbiol. 82:998-1014.
Villadangos AF, Fu HL, Gil JA, Messens J, Rosen BP, Mateos LM. (2012). Efflux permease CgAcr3-1 of Corynebacterium glutamicum is an arsenite-specific antiporter. J Biol Chem. 287:723-735.
Villadangos AF, Ordóñez E, Pedre B, Messens J, Gil JA, Mateos LM. (2014). Engineered coryneform bacteria as a bio-tool for arsenic remediation. Appl Microbiol Biotechnol. DOI 10.1007/s00253-014-6055-2.
