
Foto de grupo. De izquierda a derecha: Jon Sicilia, Jerónimo Rodríguez, Carmen de la Vega, Cristina Herencias, Álvaro San Millán, Aida Alonso, Ricardo León, Javier de la Fuente y Laura Toribio.
El grupo Plasmid Biology and Evolution (PBE lab, www.pbelab.es) nació en enero de 2016 y desde entonces ha crecido de forma sostenida hasta alcanzar su composición actual (Figura 1). El PBE lab pertenece al Área de Microbiología, Inmunología e Infección del Instituto Ramón y Cajal de Investigación Sanitaria, en el Hospital Universitario Ramón y Cajal (Madrid). En el PBE lab, nuestro principal interés es el estudio de las dinámicas evolutivas de los plásmidos en las poblaciones bacterianas. Los plásmidos son elementos genéticos que replican independientemente del cromosoma y que son capaces de transferir información genética horizontalmente entre bacterias en un proceso conocido como conjugación. De este modo, los plásmidos juegan un papel clave en la evolución y ecología bacteriana porque permiten a las bacterias acceder a nuevos caracteres adaptativos. El ejemplo más ilustrativo de la capacidad de los plásmidos para potenciar la evolución bacteriana es el papel clave que han jugado en la diseminación de mecanismos de resistencia a antibióticos. Este fenómeno ha contribuido de manera estratégica al desarrollo de la crisis global de resistencia antibiótica que afrontamos actualmente (MacLean and San Millan, 2019).
En nuestro grupo desarrollamos principalmente dos grandes líneas de investigación:
Evolución en tiempo real de la resistencia a antibióticos en el hospital
Los plásmidos conjugativos juegan un papel esencial en la diseminación de la resistencia a antibióticos en escenarios de relevancia clínica (San Millan, 2018). Uno de los factores principales que comprometen el éxito de la diseminación de los plásmidos conjugativos es el coste biológico que imponen en sus hospedadores bacterianos, lo que se traduce en una disminución de la capacidad reproductiva de las bacterias portadoras del plásmido (San Millan and MacLean, 2017). El origen de estos costes, y de la evolución compensatoria responsable de mitigarlos, continúan siendo en gran parte desconocidos. Estas dinámicas evolutivas son extremadamente relevantes, porque van a ser al menos parcialmente responsables de la aparición de las asociaciones bacteria-plásmido exitosas que se diseminan incontroladamente en los hospitales (San Millan, 2018).
En nuestro grupo intentamos comprender cuales son las bases genéticas y evolutivas que determinan el establecimiento de estas asociaciones exitosas en el hospital. Para ello, y gracias a la colaboración con los excelentes microbiólogos clínicos del hospital, estudiamos la evolución de cepas de enterobacterias portadoras de plásmidos de resistencia a antibióticos aisladas de la microbiota intestinal de pacientes hospitalizados (Figura 1). Combinamos técnicas de genómica, transcriptómica, edición genética con tecnología CRISPR, cribados CRISPRi y citometría de flujo con modelización matemática para caracterizar la epidemiología y evolución las bacterias portadoras de plásmidos de resistencia a antibióticos (DelaFuente et al., 2020; San Millan et al., 2014b; San Millan et al., 2018; San Millan et al., 2015). Nuestros resultados ayudan a predecir, y potencialmente contener, las diseminación de asociaciones bacteria-plásmido de interés clínico en nuestro hospital.
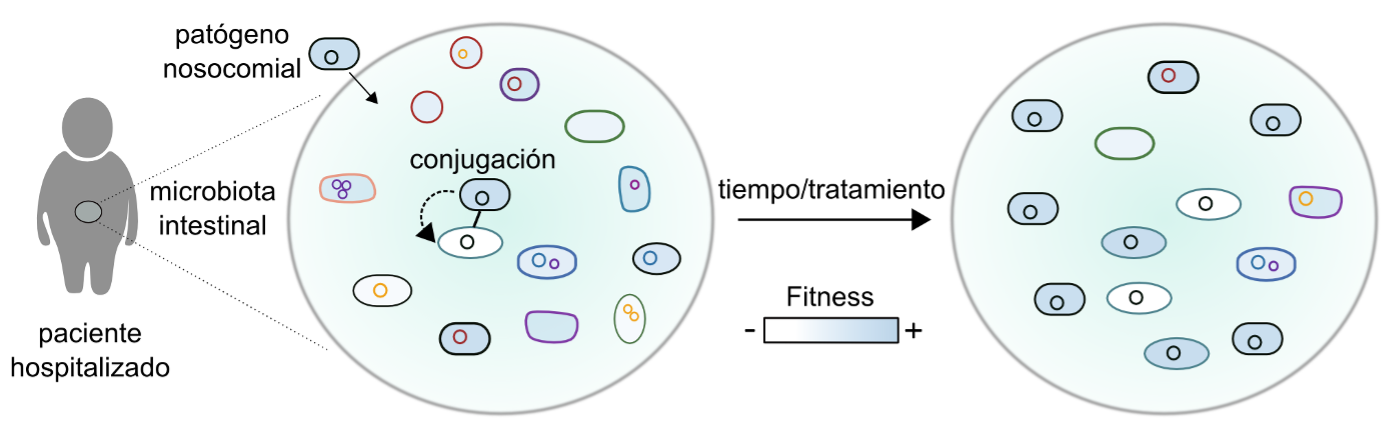
Figura 1. Evolución de la resistencia a antibióticos mediada por plásmidos. Modificada de (San Millan, 2018). Dinámicas evolutivas de asociaciones bacteria-plásmido en la microbiota intestinal de un paciente hospitalizado. Un patógeno nosocomial portador de un plásmido de resistencia a antibióticos coloniza la microbiota intestinal y transfiere el plásmido a bacterias residentes en esta microbiota. El plásmido puede producir un coste biológico (i.e. reducción de fitness. Leyenda: blanco, fitness bajo; azul, fitness alto), pero es seleccionado por un tratamiento con antibióticos. Con el tiempo, este coste desaparece gracias a mutaciones compensatorias.
Papel de los plásmidos multicopia en la evolución bacteriana
Los plásmidos se pueden dividir de manera general entre aquellos de gran tamaño y bajo numero de copias y los de pequeño tamaño y alto numero de copias. Aunque el limite de tamaño varía entre familias bacterianas, esta distribución bimodal de tamaños y copias se mantiene constante a través de la filogenia (San Millan et al., 2014a; Smillie et al., 2010). Los plásmidos conjugativos de gran tamaño juegan un papel decisivo en la diseminación de la resistencia a antibióticos y por lo tanto se han estudiado exhaustivamente durante las últimas décadas. Los plásmidos pequeños multicopia son extremadamente comunes en los procariotas, pero su papel en la biología y evolución bacteriana se ha estudiado en menor medida.
En nuestro laboratorio investigamos las posibles ventajas evolutivas asociadas a los plásmidos multicopia. Una de las características más destacadas de estos plásmidos es el hecho de que representan una isla de poliploidía en el genoma haploide de la mayoría de las bacterias. En estudios recientes, hemos demostrado que la poliploidía provista por estos plásmidos tiene al menos dos consecuencias importantes: (i) acelerar la evolución de los genes que codifican (San Millan et al., 2016) y (ii) mantener la diversidad alélica de los mismos en las bacterias hospedadoras (Rodriguez-Beltran et al., 2018) (Figura 2).
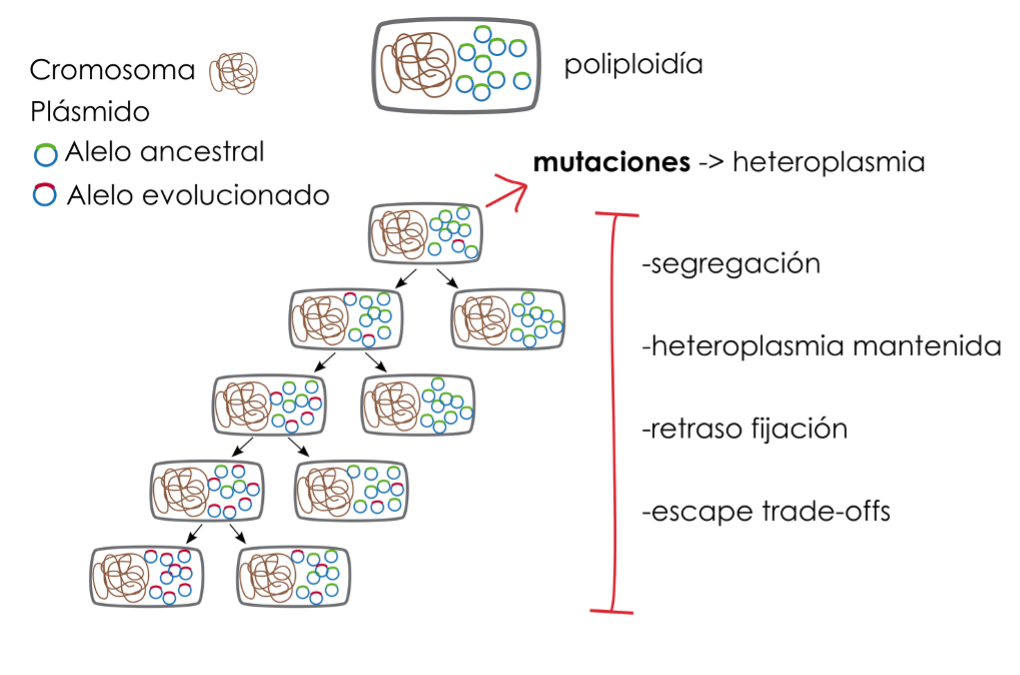
Figura 2. Dinámicas evolutivas de los plásmidos multicopia. La poliploidía asociada con los plásmidos multicopia incrementa las posibilidades de adquirir mutaciones en sus genes. La coexistencia de versiones del mismo plásmido con y sin la mutación se conoce como heteroplasmia. Las dinámicas de replicación y segregación de estos plásmidos retrasan la fijación de las mutaciones. La heteroplasmia mantenida provee a la bacteria de diversidad alélica, que puede tener un valor adaptativo permitiendo a la bacteria escapar de los desequilibrios (trade-offs) entre las distintas actividades de los alelos del gen.
En resumen, en nuestro grupo intentamos comprender la biología de poblaciones de los plásmidos de resistencia a antibióticos combinando técnicas avanzadas de biología molecular y biología evolutiva. Nuestro objetivo último es aplicar los conceptos que aprendemos del estudio de la evolución de la resistencia antibióticos mediada por plásmidos al desarrollo de estrategias mas racionales de control de las enfermedades infecciosas.
Bibliografía representativa
DelaFuente, J., Rodriguez-Beltran, J., San Millan, A., 2020. Methods to Study Fitness and Compensatory Adaptation in Plasmid-Carrying Bacteria. Methods Mol Biol 2075, 371-382.
MacLean, R.C., San Millan, A., 2019. The evolution of antibiotic resistance. Science 365, 1082-1083.
Rodriguez-Beltran, J., Hernandez-Beltran, J.C.R., DelaFuente, J., Escudero, J.A., Fuentes-Hernandez, A., MacLean, R.C., Pena-Miller, R., San Millan, A., 2018. Multicopy plasmids allow bacteria to escape from fitness trade-offs during evolutionary innovation. Nat Ecol Evol 2, 873-881.
San Millan, A., 2018. Evolution of Plasmid-Mediated Antibiotic Resistance in the Clinical Context. Trends Microbiol 26, 978-985.
San Millan, A., Escudero, J.A., Gifford, D.R., Mazel, D., MacLean, R.C., 2016. Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution 1, 0010.
San Millan, A., Heilbron, K., MacLean, R.C., 2014a. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations. ISME J 8, 601-612.
San Millan, A., MacLean, R.C., 2017. Fitness Costs of Plasmids: a Limit to Plasmid Transmission. Microbiology Spectrum 5.
San Millan, A., Peña-Miller, R., Toll-Riera, M., Halbert, Z.V., McLean, A.R., Cooper, B.S., MacLean, R.C., 2014b. Positive selection and compensatory adaptation interact to stabilize non-transmissible plasmids. Nat Commun 5, 5208.
San Millan, A., Toll-Riera, M., Qi, Q., Betts, A., Hopkinson, R.J., McCullagh, J., MacLean, R.C., 2018. Integrative analysis of fitness and metabolic effects of plasmids in Pseudomonas aeruginosa PAO1. ISME J 12, 3014-3024.
San Millan, A., Toll-Riera, M., Qi, Q., MacLean, R.C., 2015. Interactions between horizontally acquired genes create a fitness cost in Pseudomonas aeruginosa. Nat Commun 6, 6845.
Smillie, C., Garcillán-Barcia, M.P., Francia, M.V., Rocha, E.P., de la Cruz, F., 2010. Mobility of plasmids. Microbiol Mol Biol Rev 74, 434-452.
