
Foto de grupo
Candida albicans es el agente causal más frecuentemente aislado en infecciones fúngicas, pero durante las pasadas dos décadas se ha descrito un aumento continuo en los casos descritos de infecciones en mucosas y diseminadas producidas por Candida glabrata, Candida parapsilosis y Candida tropicalis. Mundialmente se considera C. glabrata como el segundo agente causal más prevalente del género Candida. El tratamiento de las infecciones causadas por C. glabrata es complicado debido a que los aislados clínicos suelen mostrar una baja susceptibilidad a las terapias antifúngicas más comunes. El establecimiento de las infecciones por C. glabrata está mediado por la pared celular del hongo que establece interacciones primarias patógeno-hospedador. Por ejemplo, la pared celular contiene adhesinas que provocan la adhesión del hongo a superficies biomédicas y a la formación de biopelículas (biofilms). Por tanto, estas adhesinas juegan un papel importante en el proceso infeccioso y debido a que proteínas similares se encuentran ausentes en células de mamífero, representan candidatos idóneos para el desarrollo de nuevos fármacos antifúngicos, vacunas anti-Candida efectivas y métodos más precisos y sistemáticos para diagnosticar las infecciones fúngicas.
Hemos descrito que el genoma de C. glabrata contiene una familia excepcionalmente amplia de genes que codifican adhesinas de la pared celular. Entre las (putativas) proteínas de tipo adhesina destacan las proteínas Epa, que representan una subfamilia de proteínas de tipo lectina. Las funciones biológicas de la mayoría de las adhesinas de pared no se conocen, si exceptuamos las de un número muy limitado de adhesinas de la subfamilia Epa. Un conocimiento más profundo de los mecanismos moleculares fundamentales que regulan la actividad de las adhesinas y su papel en la patogenia de la infección es crucial para desarrollar nuevas estrategias que permitan la prevención y el tratamiento de estas candidiasis.
Las adhesinas de la pared fúngica son proteínas modulares (Fig. 1); sus precursores contienen péptidos señal para entrar en el RE y anclajes GPI, y las proteínas maduras comprenden un dominio N-terminal, que generalmente determina la especificidad de unión a ligandos de las adhesinas seguido de un dominio de baja complejidad que en la mayoría de casos contiene repeticiones internas en tandem. Estas repeticiones internas juegan un papel importante en la adhesividad del hongo y en la exposición de los dominios de unión a ligandos modulado por el número de copias repetidas y consecuentemente, por las variaciones de tamaño de los genes que codifican adhesinas de los aislados clínicos.
Nuestros datos muestran que existen grandes variaciones fenotípicas entre los aislamientos clínicos de C. glabrata en su capacidad de adhesión a las superficies abióticas de importancia médica. Nuestra hipótesis es que estas cepas más adherentes (hiperadherentes) son mejores colonizadoras de los tejidos del huésped que las cepas de referencia y que estos fenotipos hiperadherentes son gobernados por adhesinas de la pared celular aun no caracterizadas. Por lo tanto, el objetivo principal de nuestro trabajo es identificar y caracterizar funcionalmente las adhesinas que gobiernan los fenotipos hiperadherentes de los aislamientos clínicos. Para la identificación de nuevas adhesinas utilizamos protocolos ya establecidos de identificación de proteínas de la pared celular por espectrometría de masas. Con unos de los candidatos más prometedores realizamos estudios más detallados de caracterización por clonación y expresión heteróloga, y estudios que definan sus propiedades de unión al ligando. Estudiamos, in vivo e in vitro, su relevancia en la virulencia de Candida empleando mutantes con deleciones, mediante ensayos fenotípicos. También planeamos estudiar la actividad de diferentes agentes antifúngicos sobre la capacidad de adhesión, formación de biopelícula y virulencia experimental de aislamientos clínicos hiperadherentes y de mutantes con deleción de adhesinas específicas.
Estos estudios sobre los adhesinas se llevan a cabo en colaboración con los grupos del Prof. G. Quindós (Universidad del País Vasco, Bilbao), Drs. M. Weig y O. Bader (Göttingen University, Alemania) y Dr. N. Chauhan (Rutgers University, New Jersey, USA). A través de otros estudios, el grupo de Micología Médica también colabora con grupos en la Universidad de Valencia (Prof. E. Valentin), la Universidad de Amsterdam, Holanda (Prof. C. de Koster) y la Universidad de La Serena, Chile (Prof. L. Castillo), entre otras.
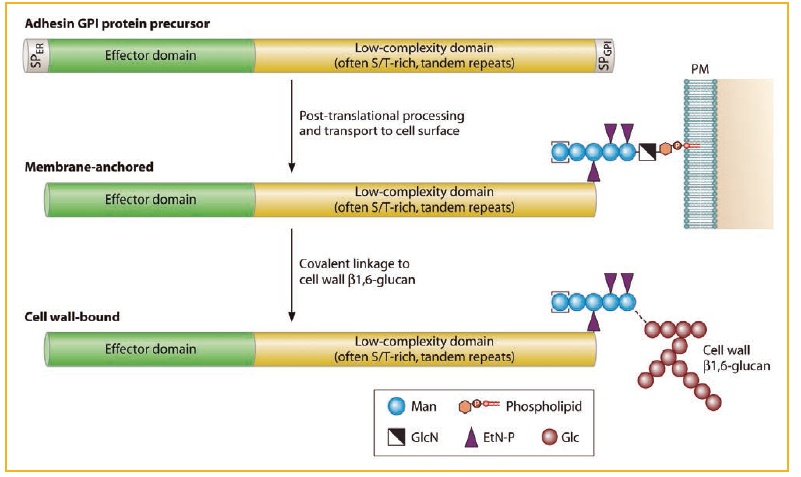
Figura 1. Estructura genérica y procesamiento post-traduccional, que lleva a la incorporacíon de adhesinas a la pared celular fúngica. No se muestra N- y O-glicosilacíon de proteínas por motivos de simplicidad.
Bibliografía representativa
Gioti A, et al., De Groot PWJ, Butler G, Heitman J, Scheynius A. (2013). Genomic Insights into the Atopic Eczema-Associated Skin Commensal Yeast Malassezia sympodialis. MBio 4: e00572-00512.
De Groot PWJ, Martinez AI, Castillo L. 2013. A genomic inventory of cell wall biosynthesis in the ubiquitous plant pathogen Botrytis cinerea. In Mora Montes HM (ed.), The Fungal Cell Wall, vol. In press. Nova Science Publishers, Inc, Hauppauge (NY).
De Groot PWJ, Bader O, De Boer AD, Weig M, Chauhan N. (2013). Adhesins in human fungal pathogens: glue with plenty of stick. Eukaryot. Cell 12: 470-481.
Wagener J, Weindl G, De Groot PWJ, De Boer AD, Kaesler S, Thavaraj S, Bader O, Mailänder-Sanchez D, Borelli C, Weig M, Biedermann T, Naglik JR, Korting HC, Schaller M. (2012). Glycosylation of Candida albicans cell wall proteins is critical for induction of innate immune responses and apoptosis of epithelial cells. PLoS ONE 7: e50518.
Thevissen K, De Mello Tavares P, Xu D, Blankenship J, Vandenbosch D, Idkowiak-Baldys J, Govaert G, Bink A, Rozental S, De Groot PWJ, Davis TR, Kumamoto CA, Vargas G, Nimrichter L, Coenye T, Mitchell A, Roemer T, Hannun YA, Cammue BPA. (2012). The plant defensin RsAFP2 induces cell wall stress, septin mislocalization and accumulation of ceramides in Candida albicans. Mol. Microbiol. 84: 166-180.
Gelis S, De Groot PWJ, Castillo L, Moragues MD, Sentandreu R, Gómez MM, Valentín E. (2012). Pga13 in Candida albicans is localized in the cell wall and influences cell surface properties, morphogenesis and virulence. Fungal Genet. Biol. 49: 322-331.
De Groot PWJ, Brandt BW. (2012). ProFASTA: A pipeline web server for fungal protein scanning with integration of cell surface prediction software. Fungal Genet. Biol. 49: 173-179.
Bader O, Schwarz A, Kraneveld EA, Tangwattanchuleeporn M, Schmidt P, Jacobsen MD, Groß U, De Groot PWJ, Weig M. (2012). Gross karyotypic and phenotypic alterations among different progenies of the Candida glabrata CBS138/ATCC2001 reference strain. PLoS ONE 7: e52218.
Laforet L, Moreno I, Sánchez-Fresneda R, Martínez-Esparza M, Martínez JP, Argüelles J-C, De Groot PWJ, Valentín E. (2011). Pga26 mediates filamentation and biofilm formation and is required for virulence in Candida albicans. FEMS Yeast Res. 11: 389–397.
Kraneveld EA, De Soet JJ, Deng DM, Dekker HL, De Koster CG, Klis FM, Crielaard W, De Groot PWJ. (2011). Identification and differential gene expression of adhesin-like wall proteins in Candida glabrata biofilms. Mycopathologia 172: 415-427.
De Boer AD, De Groot PWJ, Weindl G, Schaller M, Riedel D, Diez-Orejas R, Klis FM, De Koster CG, Dekker HL, Groß U, Bader O, Weig M. (2010). The Candida albicans cell wall protein Rhd3/Pga29 is abundant in the yeast form and contributes to virulence. Yeast 27: 61-624.
De Groot PWJ, Kraneveld EA, Yin QY, Dekker HL, Groß U, Crielaard W, De Koster CG, Klis FM, Weig M. (2008). The cell wall of the human pathogen Candida glabrata: differential incorporation of novel adhesin-like wall proteins. Euk. Cell 7: 1951-1964.
De Groot PWJ, Klis FM. (2008). The conserved PA14 domain of cell wall-associated fungal adhesins governs their glycan binding specificity. Mol. Microbiol.68: 535-537.
