
Foto de grupo. Equipo EXOPOL
EXOPOL S.L., es una spinoff de la Universidad de Zaragoza con más de 20 años de experiencia en diagnóstico y producción de autovacunas en sanidad animal; áreas a las que dedica más del 50% de su esfuerzo en I+D+i y en las que mantiene colaboraciones con más de 25 grupos de investigación a nivel nacional e internacional.A lo largo de su trayectoria, la empresa ha obtenido financiación para llevar a cabo diversos proyectos de investigación (CDTI) destinados principalmente a mejorar el diagnóstico actual y la producción de vacunas en el ámbito veterinario.
La gran experiencia de EXOPOL en estas áreas, incluido el diagnóstico molecular mediante PCR en tiempo real (qPCR), ha permitido que la empresa lleve a cabo el diseño y la validación de sus propios ensayos, a los que ha designado como “EXOone qPCR” y que cuentan con importantes ventajas comparado a la mayoría de kits comerciales de este tipo disponibles en el mercado. El ensayo EXOone qPCR, hace uso de una nueva tecnología para estabilizar todos los reactivos necesarios de la reacción en los pocillos de una placa o tiras de qPCR, lo que ha permitido obtener un producto “ready to use”, con almacenamiento a 4º C, además de otras ventajas.El estabilizado permite también almacenar estos kits de qPCR a temperaturas de refrigeración (+4°C a +8°C) sin la necesidad de congelarlos; lo que supone una gran ventaja práctica puesto que facilita el transporte y evita el tedioso proceso de descongelación de los diferentes reactivos necesarios para preparar una mezcla en un ensayo de qPCR habitual.
Otra característica importante de los ensayos EXOone qPCR es que todos han sido diseñados para utilizar el mismo protocolo térmico; lo que permite la posibilidad de evaluar en una misma carrera de amplificación varios patógenos, reduciendo costes y recursos. Esta ventaja de nuestros ensayos permite diseñar paneles específicos de diagnóstico al gusto del cliente, tal como el que se aprecia a continuación.
Actualmente, la empresa continúa diseñando nuevos productos y buscando colaboraciones con grupos de investigación interesados en aplicar esta tecnología en otras áreas del diagnóstico molecular como son salud humana, agro-alimentaria o medio ambiente entre otras .
descrito recientemente Burd et al 2010 (1) y Saunders et al 2013 (2). En EXOPOL hemos seguido estos criterios con la finalidad de brindar un producto de calidad y que sea a la vez rápido y sencillo de utilizar.
Los resultados de la validación de ensayos EXOone qPCR para la identificación de patógenos relevantes en nuestro sector como son: Coxiella burnetii (3), Leptospira patógena (4), Toxoplasma gondii (5), Mycoplasma agalactiae (6), Mycoplasma bovis (7), etc; han sido presentados en varios congresos nacionales e internacionales de gran relevancia como son el WAVLD, EAVLD, AVEDILA, SEM, entre otros. Adicionalmente, se ha realizado desarrollo de otros ensayos de qPCR para la identificación de patógenos bacterianos, parasitarios o virales de interés veterinario, cuyos resultados se encuentran actualmente en proceso de publicación (para una lista detallada visite: www.exopol.com).
La validación de los ensayos EXOone qPCR incluyen la determinación de:
– Sensibilidad analítica: El rango reportable de cuantificación fue calculado utilizando controles positivos sintéticos previamente diseñados y validados (8) para cada ensayo, los cuales han permitido determinar unos límites mínimos (LLOQ) y máximos de cuantificación (ULOQ) de entre 101 hasta 109 copias del patógeno/ reacción, con una buena linealidad (Fig 1).
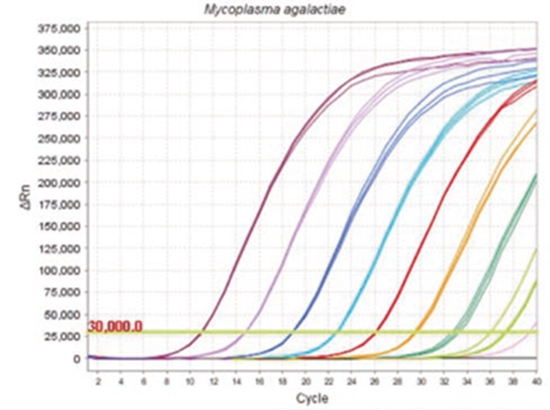
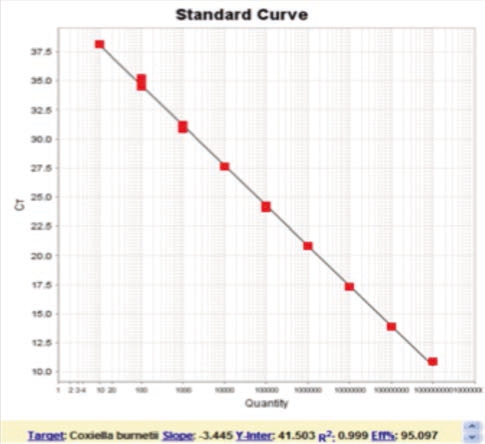
Fig.2 Estudio de intra-ensayo para el Mycoplasma agalactiae EXOone qPCR. Cada dilución del standard (CP) fue evaluada por triplicado.
– Estudios de concordancia: Mediante la evaluación de un panel de cepas de referencia, cepas vacunales, cepas de campo y casos clínicos positivos y negativos a cada patógeno; los cuales son evaluados en paralelo con los ensayos comerciales de qPCR más importantes en el mercado (LSI, Adiagen, Ingenetix, etc) para determinar el nivel de concordancia. Los ensayos EXOone qPCR han demostrado tener una excelente concordancia (Kappa 0,87 a 1.0) con los kits de qPCR ofrecidos por las empresas relevantes en nuestro sector.
– Estudios de especificidad: En un estudio que incluye la evaluación de un panel de entre 25 a 45 microorganismos (bacterias, virus y parásitos) relacionados genéticamente, que causan enfermedades relacionadas o co-infecciones, o que pueden encontrarse como microbiota natural en las muestras seleccionadas.
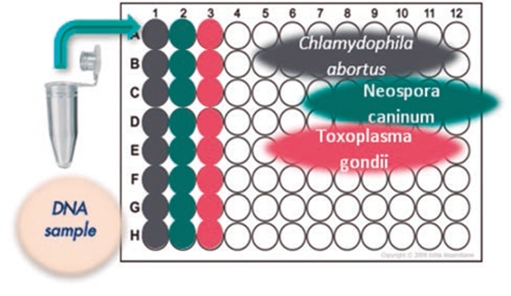
Fig.3 Panel reproductivo para la evaluación simultanea (multiparamétrico) de Chlamydophila abortus, Neospora caninum y Toxoplasma gondii.
Principales características de los ensayos EXOone qPCR
La nueva tecnología empleada en los ensayos EXOone qPCR, al estabilizar todos los reactivos necesarios en pocillos, permite una preparación más rápida de la qPCR; puesto que sólo necesita la adición de agua DEPC (suministrada con el kit) y la muestra de ácidos nucleicos, antes de llevar la reacción al termociclador. Esto hace que la labor del técnico sea mucho más sencilla y evita además los posibles errores de pipeteo durante la preparación del ensayo, con la consecuente mejora en la repetibilidad y reproducibilidad del mismo.
En resumen, el ensayo estabilizado EXOone qPCR, debido a su formato “ready to use” y su cuidada validación, son una excelente herramienta diagnóstica para la identificación o cuantificación de patógenos de interés veterinario, destacando no sólo por su sensibilidad y especificidad sino también por su facilidad de uso.
Bibliografía representativa
Benito AA, Arnal JL, de Tomas E, et al. Internal controls for reliable results in real-time pcr diagnostic assays. WAVLD, Berlin 2013.
Benito AA, Villa A, Serrano JD, et al. Desarrollo de una PCR en Tiempo Real para el diagnóstico de Toxoplasma gondii. SEM IX reunión grupo Microbiología Molecular. Mallorca, 2012.
Burd Eileen M. Validation of Laboratory-Developed Molecular Assays for Infectious Diseases. Clinical Microbiology Reviews 2010. Vol.23 (3):
Saunders N, Zambon M, Sharop I, et al. (2013). Guidance on the development and validation of diagnostic tests that depend on nucleic acid amplification and detection. Journal of Clinical Virology 56
Villa A, Arnal JL, Serrano DJ, et al. Development and validation of a Real-Time PCR Zen Gel Mix for the diagnosis and quantification of Coxiella burnetii. EAVLD, Polonia 2012a.
Villa A, Benito AA, Bernal JL et al. Leptospirosis: Diagnóstico mediante PCR en tiempo real y aislamiento microbiológico. AVEDILA, Badajoz.2012b.
Villa A, De Tomás E, Benito AA, Arnal JL, Serrano JD. Evaluation of cloned genes as quantitative controls in Real-Time assays for infectious diseases with veterinary importance. SEBBM 2012c, Sevilla.
Villa A, Fernández A, JL Arnal, et al. Desarrollo y validación de una ZEN Real Time PCR Gel Mix para la detección y cuantificación del gen (mp81) de Mycoplasma agalactiae. AVEDILA, Tenerife 2011.
Villa A, Serrano JD, Benito AA, et al. Targeting uvrC gene for Mycoplasma bovis Gel Mix Real Time PCR. WAVLD, Berlin 2013.
