
Foto de grupo. Miembros del grupo “Estructura, Dinámica y Función de Genomas de Rizobacterias” en la actualidad.
Nuestro grupo de “Estructura, Dinámica y Función de Genomas de Rizobacterias” tiene como objetivo obtener conocimientos básicos y aplicados sobre las interacciones beneficiosas entre plantas y microorganismos de interés en sistemas agrícolas y forestales (https://www.eez.csic.es/estructura-dinamica-y-funcion-de-genomas-de-rizobacterias). El grupo se constituye a finales de los años 90 como una deriva de las líneas dentro del Departamento de Microbiología del Suelo y Sistemas Simbióticos en nuestro centro – La Estación Experimental del Zaidín – basadas en el estudio de la simbiosis Rhizobium-leguminosa. El grupo está constituido por cuatro investigadores en plantilla: Nicolás Toro, Francisco Martínez-Abarca, Manuel Fernández López y Jose I. Jiménez Zurdo. Cuenta en la actualidad con un total de 14 miembros entre personal técnico, estudiantes de Máster y Predoctorales así como personal Postdoctoral. Focaliza sus líneas de investigación en la ecología de microorganismos rizosféricos y la utilización de éstos en la recuperación de suelos degradados, en el análisis genómico de comunidades microbianas del suelo, la caracterización de nuevos ARNs reguladores en microorganismos simbióticos y por último en la contribución al desarrollo de genómica funcional de microorganismos y plantas mediante el uso de herramientas de mutagénesis de alto rendimiento. Esta última línea de Investigación (@ToroLaboratory) se desarrolló partiendo de unos descubrimientos llevados a cabo en los inicios sobre un aislado particular, GR4, de la bacteria Sinorhizobium meliloti.
Durante la década de los años 80, el estudio de los determinantes genéticos causantes de una mayor eficiencia en la nodulación de plantas nos llevó a una zona concreta de 40 kb contenida en uno de los dos plásmidos crípticos presentes en este aislado (pRmeGR4b). Este punto es el comienzo de la primera deriva clave en la investigación dentro del grupo que nos llevó a interesarnos en un elemento móvil presente en esta región que ha dado lugar al desarrollo de toda una tecnología de mutagenesis a la carta derivada de lo que apenas se conocía en aquel momento (finales de los 90) como los intrones del grupo II. En esta región se encontró un locus de 1894 nt conteniendo el primer retroelemento activo en rizobacterias: RmInt1 (Rm de Rhizobium meliloti, Int1, el primer Intron). Los intrones del grupo II son grandes ribozimas que se comportan como elementos móviles que sufren “splicing” mediante un mecanismo que recuerda a los intrones eucarióticos. Su movilidad está mediada por la formación de un complejo ribonucleoproteína constituido por la proteína codificada por el propio intrón y el RNA escindido. El DNA diana hacia donde se dirige es reconocido principalmente por el RNA que puede programarse de manera eficiente hacia cualquier DNA diana a la carta.
El trabajo de sucesivos años con este elemento sentó las bases de nuevas variedades de intrones con los que desarrollar la tecnología del targetrón dando lugar a varios proyectos de investigación, así como a distintas colaboraciones internacionales. Una actividad relevante presente en este retroelemento y necesaria para su actividad como herramienta mutagénica es la actividad Reverso Transcriptasa (RT). La presencia y el éxito de este retroelemento en S. meliloti, nos ha hecho preguntarnos si su presencia en un momento determinado podría haber supuesto ventajas evolutivas a los microorganismos que lo contienen. Así, nuevos elementos relacionados, fragmentos de elementos antiguos insertados en zonas críticas conservadas en los genomas de rhizobios relacionados han aparecido en nuevos genomas secuenciados. A ello se ha unido además el esfuerzo y conocimiento del genoma completo de GR4, la bacteria de partida y que gracias a nuestro conocimiento sobre este elemento repetido nos permitió cerrarlo de manera precisa, así como abordar la secuenciación de nuevas variantes que nos ha permitido conocer los tiempos de evolución de esta bacteria y su asociación muy probable al cultivo del que dependen (en este caso la afalfa).
La nueva Microbiología, nos ha cambiado la manera de hacernos preguntas sobre el papel de intrones y otros elementos evolutivamente relacionados con ellos en los hospedadores que lo contienen. Así yendo un paso más, nos ha interesado especialmente entender el origen de la RT presente en este tipo de retroelementos, para lo que desde 2014, llevamos interesados en conocer las relaciones filogenéticas de los distintos tipos de reversos transcriptasas presentes en el mundo procariota. Ya desde ese año, observamos un subgrupo particular estrechamente relacionado con las que se encontraban presentes en los Intrones del grupo II que parecían haber cambiado completamente de sistema funcional. Estas reverso-transcriptasas se encontraban formando parte de locus CRISPR, en concreto asociados fundamentalmente a genes del complejo adaptativo, y en algunos casos formando una proteína de fusión (RT-Cas1). Los sistemas CRISPR-Cas constituyen un sistema de defensa “inmune” de las bacterias frente a virus y DNA exógeno. En la última fase del proceso, interviene una ribonucleoproteína (la conocida como cas9) capaz de reconocer “in vivo” una secuencia diana mediante un RNA guía y digerirlo de manera rápida, eficiente y precisa. Esta propiedad ha dado lugar a la denominada ‘Tecnologia CRISPR’ para la edición genómica a la carta. Una nueva pregunta surge en nuestro grupo de investigación: ¿Cual es el papel de las reversotranscriptasas en los sistemas CRISPR-Cas? ¿A qué sistemas están asociadas? ¿Qué tipo de co-evolución parecen haber tenido con respecto al sistema CRISPR-Cas al que se asocian? Nosotros y otros grupos hemos encontrado nuevas variedades de RTs estrechamente relacionadas con algunos sistemas CRISPR-Cas. En un trabajo reciente hemos estudiado un nuevo sistema RT-CRISPR-Cas funcional. En nuestro trabajo hemos demostrado el papel esencial de la actividad retrotranscriptasa en el proceso de adquisición de espaciadores. Proceso que a su vez carece de sesgos en la secuencia descritos previamente en otros sistemas. El estudio proporciona nuevas herramientas con potencial diferencial para el desarrollo de nuevas herramientas biotecnológicas.
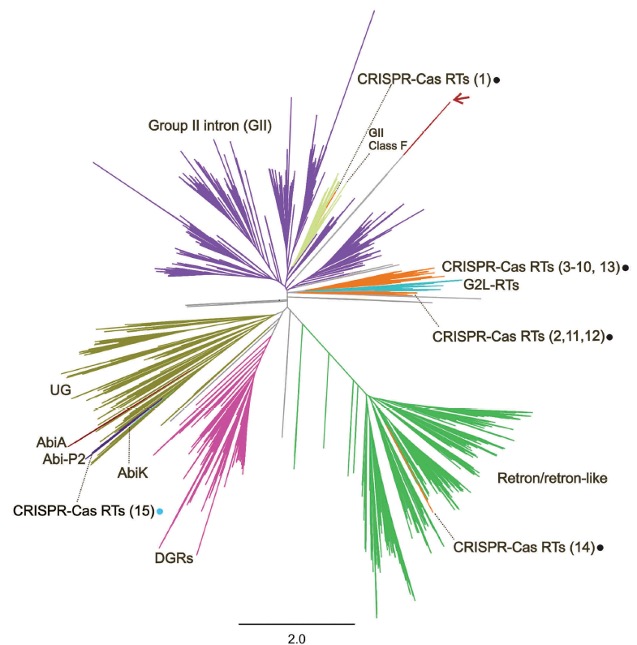
Figura 1. Arbol filogenético que representa la diversidad procariota de las Reverso Transcriptasas (RTs) identificadas hasta la fecha. Se destacan mediante puntos aquellas que se encuentran estrechamente relacionadas a sistemas CRISPR-Cas. (Modificado de Toro et al. 2019, RNA Biol.
Publicaciones recientes más relevantes
González-Delgado A, Mestre MR, Martínez-Abarca F, Toro N. (2019) Spacer acquisition from RNA mediated by a natural reverse transcriptase-Cas1 fusion protein associated with a type III-D CRISPR-Cas system in Vibrio vulnificus. Nucleic Acids Res. In press.
Toro N, Martínez-Abarca F, Mestre MR, González-Delgado A. (2019) Multiple origins of reverse transcriptases linked to CRISPR-Cas systems. RNA Biol. 16:1486-1493.
García-Rodríguez FM, Neira JL, Marcia M, Molina-Sánchez MD, Toro N. (2019) A group II intron-encoded protein interacts with the cellular replicative machinery through the β-sliding clamp. Nucleic Acids Res. 47:7605-7617.
Toro N, Martínez-Abarca F, Molina-Sánchez MD, García-Rodríguez FM, Nisa-Martínez R. (2018) Contribution of Mobile Group II Introns to Sinorhizobium meliloti Genome Evolution. Front Microbiol. 9:627.
Toro N, Martínez-Abarca F, González-Delgado A. (2017) The Reverse Transcriptases Associated with CRISPR-Cas Systems. Sci Rep. 7(1):7089.
Toro N, Martínez-Abarca F, Fernández-López M. (2016) The early events underlying genome evolution in a localized Sinorhizobium meliloti population. BMC Genomics.17:556.
Toro N, Nisa-Martínez R. (2014) Comprehensive phylogenetic analysis of bacterial reverse transcriptases. PLoS One. 9(11):e114083.
Toro N, Martínez-Rodríguez L, Martínez-Abarca F. (2014) Insights into the history of a bacterial group II intron remnant from the genomes of the nitrogen-fixing symbionts Sinorhizobium meliloti and Sinorhizobium medicae. Heredity (Edinb).113(4):306-15.
García-Rodríguez FM, Hernández-Gutiérrez T, Díaz-Prado V, Toro N. (2014) Use of the computer-retargeted group II intron RmInt1 of Sinorhizobium meliloti for gene targeting. RNA Biol. 11(4):391-401.
Martínez-Abarca F, Martínez-Rodríguez L, López-Contreras JA, Jiménez-Zurdo JI, Toro N. (2013) Complete Genome Sequence of the Alfalfa Symbiont Sinorhizobium/Ensifer meliloti Strain GR4. Genome Announc. 1(1). pii: e00174-12.
