
Foto de grupo. Laboratorio para el Estudio del Microbioma Humano, integrado por Luis Alcaraz (actualmente en la UNAM, México), Pedro Belda, Álex Mira, Áurea Simón, Raúl Cabrera (de izquierda a derecha en la foto principal). Anny Camelo, Arantxa López, Alba Boix, Mariam Ferrer y César Bernabé (fotos inferiores, de izquierda a derecha).
El Laboratorio para el Estudio del Microbioma Humano está dirigido por el Dr Alex Mira Obrador, y se creó en el año 2009, al integrarse en el Área de Genómica y Salud de la Fundación FISABIO. La investigación del grupo se centra en caracterizar la microbiota humana comensal y patógena desde una aproximación mixta de bioinformática y trabajo experimental en genómica y metagenómica. Se trata de un equipo multidisciplinar que incluye microbiólogos, biólogos moleculares, dentistas, farmacéuticos y bioinformáticos.
(Foto de grupo). El Dr Mira ha sido uno de los fundadores de la Red Nacional de Genómica Bacteriana. Su contribución científica a este campo le valió en el año 2009 el Premio Jaime Ferrán de la Sociedad Española de Microbiología. El grupo está integrado por tres estudiantes predoctorales (dos de ellos en su fase final de tesis), dos investigadores postdoctorales y una estudiante de Máster y cuenta cada año con 2-4 estudiantes en prácticas nacionales e internacionales. La formación que se ofrece en el grupo es una aproximación mixta de técnicas experimentales y computacionales. El Laboratorio tiene reuniones semanales de gestión y discusión de artículos científicos, y realiza un curso anual de análisis masivo de secuencias para estudiar biodiversidad.
Microbioma y diagnóstico de enfermedades
Hemos utilizado técnicas de secuenciación masiva para identificar posibles microorganismos asociados al cáncer colorrectal. Las biopsias de tumores y pólipos mostraron una microbiota claramente distinta de aquella localizada en zonas sanas del colon (Mira-Pascual et al. 2014), lo cual es muy prometedor para utilizar la comunidad microbiana como biomarcadores del cáncer antes de la aparición de signos clínicos, lo cual ha sido patentado recientemente en colaboración con la empresa Entrechem SL.
En cuanto a la caries dental, hemos determinado que las bacterias presentes en la saliva no son buenos marcadores de esta patología. Sin embargo, hemos identificado una serie de metabolitos que cuando se miden en este fluido, nos indican con gran precisión si la persona tiene caries. Este descubrimiento ha sido patentado como test de diagnóstico del riesgo de caries, y al indicar la causa del riesgo, permitirá dar un tratamiento preventivo personalizado a cada paciente.
Formación de biopelículas en tiempo real
El grupo ha desarrollado un nuevo modelo de biofilm in vitro donde se puede monitorizar el crecimiento de biopelículas monoespecíficas y complejas (como la placa dental) en tiempo real. Este modelo de biofilm está siendo utilizado para identificar moléculas inhibidoras del Quórum Sensing y para medir el crecimiento de las bacterias que causan infecciones en catéteres e implantes médicos en tiempo real, identificando no sólo las dosis inhibitorias necesarias sino también que algunos antibióticos de hecho estimulan la producción de más biofilm. Todo ello supondrá un enorme. ahorro económico en el sistema de salud, pues se podría ver la resistencia a antibióticos de las bacterias causantes de infecciones post-operatorias y decidir un tratamiento efectivo en la mitad de tiempo actual.
La leche contiene microorganismos
El grupo ha aplicado las técnicas de secuenciación masiva para descifrar la enorme diversidad de bacterias que contiene el calostro y la leche materna (Cabrera-Rubio et al. 2012), trabajo que puede tener una enorme trascendencia en el campo de la nutrición infantil. Hemos encontrado estas bacterias en muestras fecales del bebé, por lo que consideramos que la leche puede ser la solución que la evolución humana ha encontrado para una transmisión controlada de los microorganismos de una generación a la siguiente. La función de la microbiota de la leche materna está aún por determinar, y aunque no podemos descartar una función metabólica, trabajamos con la hipótesis de que estas bacterias sean fundamentales para el correcto desarrollo del sistema inmune del bebé, incidiendo en enfermedades como el asma o las alergias, que epidemiológicamente se encuentran en mayores proporciones en niños alimentados con leche de fórmula.
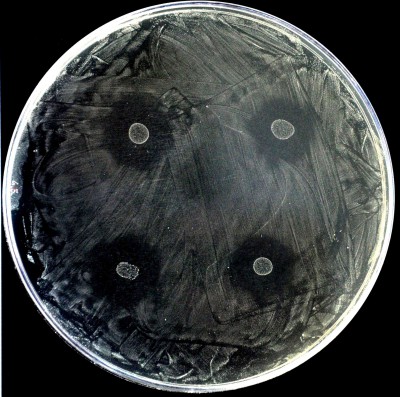
Figura. Cultivo en tapiz de la bacteria causante de la caries Streptococcus mutans, que se ve inhibida por las bacteriocinas producidas por cuatro colonias de Streptococcus dentisani, actualmente desarrollado como probiótico anti-caries.
Otros microbiomas
Las vías bajas respiratorias, tradicionalmente consideradas estériles, contienen una enorme diversidad de microorganismos, al menos en pacientes con Enfermedad Pulmonar Obstructiva Crónica, y esta población bacteriana varía dependiendo de los episodios de estabilidad o crisis del paciente (Galiana et al. 2014). También hemos determinado que el esputo, muestra de elección en la mayoría de estudios clínicos por ser no invasiva, no es en absoluto representativa de la diversidad microbiana en la mucosa pulmonar (Cabrera-Rubio 2012b). También hemos colaborado con el IPLA-CSIC y con el Hospital de Elche en estudios de la microbiota del estómago mediante pirosecuenciación, mostrando que éste goza de una enorme diversidad, sobre todo en ausencia de Helicobacter pylori (Delgado et al. 2013), bacteria en la que además estamos interesados por su posible implicación en enfermedades autoinmunes.
Genómica bacteriana
El trabajo más tradicional del grupo estaba centrado en la genómica comparada y evolutiva microbiana y, aunque ya no es nuestra principal línea de investigación, seguimos realizando trabajos en este campo. Por ejemplo, en el concepto del pan-genoma bacteriano (Mira et al. 2010), en la identificación de islas de patogénesis mediante reclutamiento metagenómico (Belda-Ferre et al. 2011) o en un nuevo método para la caracterización de orígenes de replicación en procariotas. Mediante esta aproximación, basada en la secuenciación masiva, hemos identificado cuatro orígenes de replicación en la arquea Pyrobaculum (Pelve et al. 2012).
Bibliografía representativa
Belda-Ferre P, Cabrera-Rubio R, Moya A, Mira A. (2011). Mining virulence genes using metagenomics. PLoS One 6(10):e24975.
Belda-Ferre P, Alcaraz LD, Cabrera-Rubio R, Romero H, Simón-Soro A, Pignatelli M, Mira A. (2012). The oral metagenome in health and disease. ISME J. 6(1):46-56.
Benítez-Páez A, Belda-Ferre P, Simón-Soro A, Mira A. (2014). Microbiota diversity and gene expression dynamics in human oral biofilms. BMC Genomics. 15:311.
Cabrera-Rubio R, Collado MC, Laitinen K, Salminen S, Isolauri E, Mira_A. (2012a). The human milk microbiome changes over lactation and is shaped by maternal weight and mode of delivery. Am J
Clin Nutr. 96(3):544-51.
Cabrera-Rubio R, Garcia-Núñez M, Setó L, Antó JM, Moya A, Monsó E, Mira A. (2012b). Microbiome diversity in the bronchial tracts of patients with chronic obstructive pulmonary disease. J Clin Microbiol.
50(11):3562-8.
Camelo-Castillo A, Benítez-Páez A, Belda-Ferre P, Cabrera-Rubio R, Mira A. (2014). Streptococcus dentisani sp. nov., a novel member of the mitis group. Int J Syst Evol Microbiol. 64(Pt 1):60-5.
Delgado S, Cabrera-Rubio R, Mira A, Suárez A, Mayo B. (2013). Microbiological survey of the human gastric ecosystem using culturing and pyrosequencing methods. Microb Ecol. 65(3):763-72.
Galiana A, Aguirre E, Rodriguez JC, Mira A, Santibañez M, Candela I, Llavero J, Garcinuño P, López F, Ruiz M, Garcia-Pachon E, Royo G. (2014). Sputum microbiota in moderate versus severe patients with COPD. Eur Respir J. 43(6):1787-90.
Mira A, Martín-Cuadrado AB, D’Auria G, Rodríguez-Valera F. (2010). The bacterial pan-genome: a new paradigm in microbiology. Int Microbiol. 13(2):45-57.
Mira-Pascual L, Cabrera-Rubio R, Ocon S, Costales P, Parra A, Suarez A, Moris F, Rodrigo L, Mira A, y Collado MC. (2014). Microbial mucosal colonic shifts associated with the development of colorectal cancer reveal the presence of different bacterial and archaeal biomarkers. J Gastroenterol.
Pelve EA, Lindås AC, Knöppel A, Mira A, Bernander R. (2012). Four chromosome replication origins in the archaeon Pyrobaculum calidifontis. Mol Microbiol. 85(5):986-95.
Simón-Soro A, Belda-Ferre P, Cabrera-Rubio R, Alcaraz LD, Mira A. (2013). A tissue-dependent hypothesis of dental caries. Caries Res. 47(6):591-600.
Simón-Soro A, Guillen-Navarro M, y Mira A. (2014). Metatranscriptomics reveals overall active bacterial composition in caries lesions. J Oral Microbiol 6:25443.
