
Foto de grupo. De izquierda a derecha. Agachados: Igor Ruiz de los Mozos, Begoña García, Maite Villanueva. De pie: Saioa Burgui,Iñigo Lasa, Tana Taglialegna, Sonia Saenz, Carlos Caballero, Maite Villanueva, Cristina Solano, Jaione Valle, Alejandro Toledo, Amaia Sabalza y Juani Prieto.
Al igual que el hombre, las bacterias prefieren vivir en urbes donde la compañía de otras bacterias facilita el desarrollo de una matriz en cuyo interior las bacterias viven protegidas de cambios en las condiciones ambientales y en un entorno donde es mas sencillo acumular enzimas degradativas, retener agua o comunicarse. El grupo de Biofilms bacterianos inició su andadura en 1999 y desarrolla su actividad investigadora en el Instituto de Agrobiotecnología, un centro mixto de investigación entre la Universidad Pública de Navarra, el CSIC y el Gobierno de Navarra.Su investigación se centra en las bases moleculares y genéticas del proceso de formación del biofilm de dos bacterias patógenas: Staphylococcus aureus y Salmonella enteritidis ser. Enteritidis. La razón que nos llevo a elegir bacterias filogenéticamente tan alejadas para realizar nuestros estudios fue el convencimiento de que esta estrategia nos permitiría reconocer aquellos elementos que son comunes al proceso de formación del biofilm de aquellos otros elementos que son específicos para cada bacteria. Staphylococcus aureus es una de las bacterias patógenas que mayores problemas sanitarios causa en los países desarrollados. Esto se debe a (i) la enorme cantidad de factores de virulencia que es capaz de producir y que le proporcionan una gran versatilidad como patógeno; (ii) la existencia de cepas multirresistentes a los antibióticos (las comúnmente denominadas MRSA); y (iii) a su capacidad para adherirse a los implantes médicos y causar infecciones mediadas por biofilms. Esta última característica fue determinante a la hora de decidirnos a utilizarla como modelo de estudio del proceso de formación del biofilm. A lo largo de estos años hemos aprendido que la matriz del biofilm de S. aureus puede ser de naturaleza proteica (previamente, existía la idea de que la matriz del biofilm es siempre de naturaleza polisacarídica) y que en la regulación de la síntesis del exopolisacárido interviene una compleja cascada de reguladores cuyas conexiones todavía no entendemos bien (Toledo-Arana et al., 2001; Valle et al., 2003; Toledo-Arana et al., 2005; Valle et al., 2007; Merino et al., 2009; Vergara-Irigarayet al., 2009; Valle et al., 2011; Gil et al., 2014). También hemos estudiado los sistemas de transducción de señal de dos-componentes que conectan los estímulos ambientales con la maquinaria de síntesis de la matriz. Mas recientemente,utilizando metodologías de análisis transcriptómico hemos descubierto la existencia de un proceso global de transcripción solapante en bacterias Gram positivas. Según nuestros resultados, que han sido confirmados posteriormente por otros autores, los RNAs solapantes son digeridos por una RNasa especifica de RNA de cadena doble,RNase III, a fragmentos de 19-21 nucleótidos (Lasa et al., 2011; Lybecker et al., 2014). Este proceso de digestión de los RNAs solapantes tiene lugar a lo largo de todo el genoma y proporciona un método muy sencillo para coordinarla expresión de los genes contiguos(Lasa et al., 2012; Sesto et al., 2013; Lasa and Villanueva, 2014). El análisis transcriptómico también nos ha permitido encontrar la existencia de largas regiones no traducidas en el extremo 3’ (3’ UTR) de muchos genes de S. aureus. Estudios dirigidos a investigar la función de la 3’ UTR utilizando como modelo de estudio la región 3’ UTR del gen icaR, un regulador de la síntesis del principal exopolisacárido de la matriz del biofilm, han mostrado que las 3’ UTRs bacterianas también pueden contener elementos reguladores de la expresión génica (Ruiz de Los Mozos et al., 2013).
En el caso de Salmonella, nuestros estudios inicialmente se centraron en identificar elementos de la matriz del biofilm, donde contribuimos con la identificación de los operones responsables de la síntesis de celulosa y de una proteína de superficie con homología a una proteína de S. aureus (Solano et al., 2002; Latasa et al., 2005). Posteriormente nos focalizamos en el sistema de transducción de señal mediado por c-di-GMP donde realizamos un original abordaje que consistió en la eliminación de los elementos de esta ruta de transducción de señal de esta bacteria(García et al., 2004; Solano et al., 2009; Zorraquinoet al., 2013).
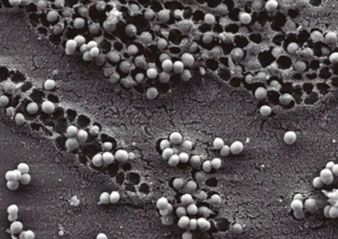
Figura 1. Biofilm de bacterias de Staphylococcus aureus
La utilización de esta cepa nos está permitiendo investigar los procesos regulados por el c-di-GMP en esta bacteria y averiguar los mecanismos de especificidad que permiten al c-di-GMP producido ante un determinado estímulo influir específicamente en sus dianas, sin interferir con las dianas activadas por otros estímulos ambientales.Aunque la motivación principal de nuestra investigación es responder a preguntas básicas de cuando, como, y para qué las bacterias forman los biofilm, siempre intentamos identificar aplicaciones biotecnológicas en nuestros resultados. Fruto de esa vocación, el grupo cuenta con dos patentes internacionales en explotación y varios miembros del grupo han constituido una empresa de base tecnológica dedicada al diseño y construcción de microorganismos modificados genéticamente “a la carte”. Nuestro grupo mantiene colaboraciones con muchos grupos para el desarrollo de sus investigaciones. Mantenemos una larga y estrecha colaboración con el grupo del Dr. José R. Penadés, anteriormente en Segorbe, pero que recientemente se ha desplazado a la Universidad de Glasgow. También mantenemos colaboraciones para la caracterización de las proteínas de la matriz del biofilm con el grupo del Dr. Jean Marc Ghigo (Instituto Pasteur), el grupo de la Dra. P. Romby (Universidad de Estrasburgo), la Dra. Cecilia Arraiano (Universidad de Lisboa) y el Dr. F. Vandenesh (Universidad de Lyon) para el análisis del proceso de transcripción solapante. En los aspectos de virulencia y genética de Salmonella, siempre hemos contado con el consejo y la ayuda de los grupos del Dr. F. García del Portillo (CNB, Madrid) y el Dr. J. Casadesus (Universidad de Sevilla).
Bibliografía representativa
García, B., Latasa, C., Solano, C., García-del Portillo, F., Gamazo, C., and Lasa, I. (2004). Role of the GGDEF protein family in Salmonella cellulose biosynthesis and biofilm formation. Mol Microbiol 54: 264–277.
Gil, C., Solano, C., Burgui, S., Latasa, C., García, B., Toledo-Arana, A., et al.(2014). Biofilm Matrix Exoproteins Induce a Protective Immune Response against Staphylococcus aureus Biofilm Infection. Infect Immun 82: 1017–1029.
Lasa, I., and Villanueva, M. (2014). Overlapping transcription and bacterial RNA removal. PNAS111: 2868–2869.
Lasa, I., Toledo-Arana, A., and Gingeras, T. (2012). An effort to make sense of antisense transcription in bacteria. RNA biology 9: 1039–1044.
Lasa, I., Toledo-Arana, A., Dobin, A., Villanueva, M., de Los Mozos, I.R., Vergara-Irigaray, M., et al. (2011). Genome-wide antisense transcription drives mRNA processing in bacteria. Proc Natl Acad Sci USA 108: 20172–20177.
Latasa, C., Roux, A., Toledo-Arana, A., Ghigo, J.-M., Gamazo, C., Penadés, J.R., and Lasa, I. (2005). BapA, a large secreted protein required for biofilm formation and host colonization of Salmonella enterica serovar Enteritidis. Mol Microbiol 58: 1322–1339.
Lybecker, M., Zimmermann, B., Bilusic, I., Tukhtubaeva, N., and Schroeder, R. (2014). The double-stranded transcriptome of Escherichia coli. Proc Natl Acad Sci USA 111: 3134–3139.
Merino, N., Toledo-Arana, A., Vergara-Irigaray, M., Valle, J., Solano, C., Calvo, E., et al.(2009). Protein A-mediated multicellular behavior in Staphylococcus aureus. J Bacteriol 191: 832–843.
Ruiz de Los Mozos, I., de Los Mozos, I.R., Vergara-Irigaray, M., Segura, V., Villanueva, M., Bitarte, N., et al.(2013). Base pairing interaction between 5“- and 3-”UTRs controls icaR mRNA translation in Staphylococcus aureus. PLoSGenetics 9: e1004001–e1004001.
Sesto, N., Wurtzel, O., Archambaud, C., Sorek, R., and Cossart, P. (2013). The excludon: a new concept in bacterial antisense RNA-mediated gene regulation. NatRev Micro 11: 75–82.
Solano, C., García, B., Latasa, C., Toledo-Arana, A., Zorraquino, V., Valle, J., et al.(2009). Genetic reductionist approac hfor dissecting individual roles of GGDEF proteins within the c-di-GMP signaling network in Salmonella. Proc Natl Acad Sci USA 106: 7997–8002.
Solano, C., García, B., Valle, J., Berasain, C., Ghigo, J.-M., Gamazo, C., and Lasa, I. (2002). Genetic analysis of Salmonella enteritidis biofilm formation: critical role of cellulose. Mol Microbiol 43: 793–808.
Toledo-Arana, A., Merino, N., Vergara-Irigaray, M., Débarbouillé, M., Penadés, J.R., and Lasa, I. (2005). Staphylococcus aureus develops an alternative, ica-independent biofilm in the absence of the arlRS two-component system. J Bacteriol 187: 5318–5329.
Toledo-Arana, A., Valle, J., Solano, C., Arrizubieta, M.J., Cucarella, C., Lamata, M., et al.(2001). The Enterococcal Surface Protein, Esp, Is Involved in Enterococcus faecalis Biofilm Formation. Appl Environ Microbiol 67: 4538–4545.
Valle, J., Latasa, C., Gil, C., Toledo-Arana, A., Solano, C., Penadés, J.R., and Lasa, I. (2011). Bap, a biofilm matrix protein of Staphylococcus aureus prevents cellular internalization through binding to GP96 host receptor. PLoSPathog 8: e1002843–e1002843.
Valle, J., Toledo-Arana, A., Berasain, C., Ghigo, J.-M., Amorena, B., Penadés, J.R., and Lasa, I. (2003). SarA and not sigmaB is essential for biofilm development by Staphylococcus aureus. Mol Microbiol 48: 1075–1087.
Valle, J., Vergara-Irigaray, M., Merino, N., Penadés, J.R., and Lasa, I. (2007). sigmaB regulates IS256-mediated Staphylococcus aureus biofilm phenotypic variation. J Bacteriol 189: 2886–2896.
Vergara-Irigaray, M., Valle, J., Merino, N., Latasa, C., Garcia, B., Ruiz de los Mozos, I., et al.(2009). Relevant role of fibronectin-binding proteins in Staphylococcus aureus biofilm-associated foreign-body infections. Infect Immun 77: 3978–3991.
Zorraquino, V., García, B., Latasa, C., Echeverz, M., Toledo-Arana, A., Valle, J., et al. (2013). Coordinated cyclic-di-GMP repression of Salmonella motility through YcgR and cellulose. J Bacteriol 195: 417–428.
