Foto de grupo: Laura Álvarez, Sara B. Hernández, Akbar Espaillat, Felipe Cava, OskarForsmo, Carlos Terriente, Teresa del Peso-Santos, Eleonore Skärfstad, Emilie Nordström y Alena Aliashkevich.
El grupo de Biología y Genética de la Pared Bacteriana (http://www.mims.umu.se/groups/felipe-cava.html) es un grupo joven que se inició en el CBMSO (CSIC-UAM) de Madrid en 2011, y hace tan solo un año nos trasladamos a Umeå, Suecia. Nuestro grupo forma parte del Departamento de Biología Molecular de la Universidad de Umeå, del MIMS-EMBL (Laboratory of Molecular Infection Medicine Sweden) y UCMR (Umeå Centre for Microbial Research). MIMS y UCMR proporcionan un ambiente interdisciplinar dotado de tecnologías de vanguardia en microscopia, metabolómica, NMR y análisis bioinformático avanzado
Nuestro principal interés es el estudio de uno de los principales “talones de Aquiles” de las bacterias: la pared celular. Estudiamos cómo las bacterias modulan su pared para adaptarse a cambios ambientales y a las distintas etapas del proceso infectivo. Para ello hacemos uso de una gran variedad de técnicas analíticas avanzadas, microscopia de correlación y análisis de imagen, combinadas con genética molecular, bioquímica, bioinformática y biología molecular. Nuestros hallazgos permitirán dar respuestas a aspectos fundamentales en fisiología bacteriana así como desarrollar nuevas estrategias para combatir enfermedades infecciosas emergentes.
Buscamos gente entusiasta que quiera unirse a nuestro grupo como postdoc. Os animamos a que nos enviéis vuestro currículo y una carta de presentación.
Nuestros proyectos
1- Biología de los D-aminoácidos
En trabajos anteriores hemos descrito el papel de los D-aminoácidos como moduladores del metabolismo de la pared celular y su función como moléculas de señalización/comunicación (17). Aunque inicialmente detectamos los D-aminoácidos no canónicos (NCDAA) en los cultivos de Vibrio cholerae, el patógeno causante del cólera, en realidad, una gran variedad de bacterias no necesariamente relacionadas taxonómicamente pueden liberar altas concentraciones de NCDAA al medio extracelular. Su acumulación coincide además con la transición a la fase estacionaria y regula negativamente la síntesis de la pared bacteriana, por lo que los NCDAA podrían estar implicados en coordinar el metabolismo de la pared celular y el citoplasma cuando los nutrientes escasean. Dado que los NCDAA parecen ser un rasgo común de diversas bacterias y que disponemos de una extensa colección (más de 1000 especies diferentes), actualmente estamos investigando su papel en diversos modelos bacterianos y en diferentes procesos fisiológicos tales como la esporulación, la formación del biofilm y la producción de metabolitos secundarios.
Además hemos caracterizado las rutas de incorporación de los NCDAA (16), lo cual tiene gran impacto en comunidades polimicrobianas, puesto que bacterias incapaces de producir estas moléculas son sin embargo capaces de incorporarlas a su pared celular. Recientemente hemos llevado a cabo una caracterización bioquímica y funcional de una familia entera de racemasas de amplio espectro, las enzimas multiespecíficas responsables de la producción de NCDAA en bacterias (8).
2- Nuevos modelos de crecimiento de pared celular en bacteria
Basándonos en la capacidad de las bacterias de incorporar diversos tipos de D-aminoácidos, hemos diseñado estrategias para el seguimiento del crecimiento y dinámica de la pared celular in vivo mediante la utilización de D-aminoácidos fluorescentes y química bioortogonal (12, 13). La universalidad de este método ha revolucionado la forma de realizar estudios del crecimiento y dinámica de la pared celular en el presente (PLoS Biol. 2013 Dec 11(12):e1001728; Nature 2013 Dec 11, NatCommun 2013 4:2856). Empleando técnicas avanzadas como la microscopía de correlación, llevamos a cabo un seguimiento espacio-temporal de la dinámica de remodelado del peptidoglicano en células vivas.
3- Caracterización de la diversidad de la mureína en el reino Bacteria
Recientemente, hemos puesto a punto la tecnología UPLC-MS en línea para el análisis “highthrough-put” del peptidoglicano que nos permitirá analizar miles de microbios en diversas condiciones. Se trata de programa de investigación muy ambicioso que pretende descubrir y explotar la variabilidad, tanto diversidad como plasticidad, de la pared celular bacteriana. Esta investigación es crítica para poder comprender la biología de la pared celular en la naturaleza, la relación de las bacterias con otros organismos y su capacidad de adaptación a desafíos medioambientales. Con los datos obtenidos estamos creando la primera base de datos de la pared celular bacteriana: el MUREINOMA. Esta enciclopedia de la pared bacteriana contribuirá al descubrimiento de nuevas rutas metabólicas y de regulación de gran interés en el desarrollo de nuevas terapias antimicrobianas más específicas.
4- Búsqueda de nuevos metabolitos moduladores de la pared bacteriana.
Otra de nuestras líneas de investigación consiste en estudiar el impacto de metabolitos ambientales en la pared celular mediante el uso tanto de librerías de compuestos químicos como de extractos biológicos más complejos (extractos marinos producidos por Actinobacterias procedentes del Ártico).
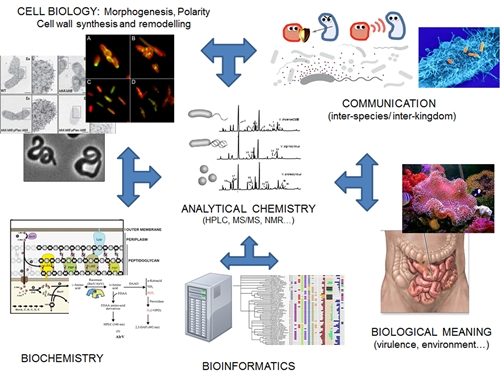
Figura 1. Esquema de las actividades del grupo de Biología y Genética de la Pared Bacteriana. En nuestro laboratorio llevamos a cabo un esfuerzo multidisciplinar para comprender el valor adaptativo/fisiológico que tiene para la bacteria el control sobre la composición y estructura de su pared.
5- Desarrollo y dispersión del biofilm de Vibrio cholerae. La producción de matriz extracelular es una característica común de las comunidades multicelulares, aunque existe gran diversidad en su formación. Estudios recientes sugieren que algunas moléculas de señalización ejercen un papel importante en el desarrollo del biofilm. Sin embargo, los mecanismos moleculares responsables de la conexión entre la comunicación química y la arquitectura del biofilm en comunidades polimicrobianas continúa siendo un misterio.Nuestro laboratorio utiliza la tecnología de RNA seq para descifrar mecanismos moleculares mediante los cuales ciertos estímulos ambientales gobiernan el desarrollo del biofilm de Vibrio cholerae. En el campus trabajan investigadores de gran prestigio internacional dentro de las áreas de microbiología molecular y biología de la infección. Uno de los objetivos principales de nuestro grupo es formar a la siguiente generación de científicos mediante la creación de un ambiente estimulante que impulse el aprendizaje a través de la colaboración.
Bibliografía representativa
Moll A, Dörr T, Alvarez L, Chao MC, Davis BM, Cava F y Waldor MK. (2014). Cell separation in Vibrio cholerae is mediated by a single amidase whose action is modulated by two non-redundant activators. J Bacteriol. In press
Alvarez L, Espaillat A, Hermoso J A, de Pedro M A y Cava F. (2014). Peptidoglycan Remodeling by the Coordinated Action of Multispecific Enzymes. Microb Drug Resist.20(3):190-8
Hernández SB, Cava F, Pucciarelli MG, García-Del Portillo F, de Pedro MA, Casadesús J. (2014). Bile-induced peptidoglycan remodeling in Salmonella enterica. Environ Microbiol. doi: 10.1111/1462-2920.12491
Cava F y de Pedro MA. (2014). Peptidoglycan plasticity in bacteria: emerging variability of the mureinsacculus and their associated biological functions. CurrOpinMicrobiol; 18C:46-53.
Dörr T, Lam H, Alvarez L, Cava F, Davis BM y Waldor MK. (2014). A Novel Peptidoglycan Binding Protein Crucial for PBP1A-Mediated Cell Wall Biogenesis in Vibrio cholerae. PLoSGenet. Jun 19;10(6):e1004433.
Dörr T, Möll A, Chao MC, Cava F, Lam H, Davis BM y Waldor MK. (2014). Differential requirement for PBP1a and PBP1b in the in vivo and in vitro fitness of Vibrio cholerae. InfectImmun. 82(5):2115-24.
Desmarais SM, Cava F, De Pedro MA y Huang KC. (2014). Isolation and preparation of bacterial cell walls for compositional analysis by ultra performance liquid chromatography. J Vis Exp. (83). doi: 10.3791/51183.
Espaillat A, Carrasco-Lopez C, Bernardo-Garcia N, Pietrosemolli N, Otero L H, Alvarez L, de Pedro M A, Pazos F, Davis B M, Waldor M K, Hermoso J A y Cava F. (2014). Structural basis for the broad specificity of a new family of amino acid racemases. Acta Crystallogr D BiolCrystallogr, 70:79-90
Cava F, Kuru E, Brun YV y de Pedro MA. (2013). Modes of cell wall growth differentiation in rod-shaped bacteria. CurrOpinMicrobiol. 16(6):731-7
Dorr T, Cava F, Lam H, Davis BM y Waldor MK (2013). Substrate specificity of an elongation-specific peptidoglycan endopeptidase and its implications for cell wall architecture and growth of Vibrio cholerae. MolMicrobiol, Jul 9, doi: 10.1111/mmi.12323.
Desmarais SM, De Pedro MA, Cava F y Huang KC (2013). Peptidoglycan at its peaks: how chromatographic analyses can reveal bacterial cell wall structure and assembly. MolMicrobiol 89 (1): 1-13.
Siegrist MS, Whiteside S, Jewett JC, Aditham A, Cava F y Bertozzi CR (2013). (D)-amino acid chemical reporters reveal peptidoglycan dynamics of an intracellular pathogen. ACS ChemBiol 8 (3): 500-505.
Kuru E, Hughes HV, Brown PJ, Hall E, Tekkam S, Cava F, de Pedro MA, Brun YV y VanNieuwenhze MS (2012). In situ probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids.AngewChemInt Ed Engl 51 (50): 12519-12523.
Horcajo P, de Pedro MA y Cava F (2012). Peptidoglycan plasticity in bacteria: stress-induced peptidoglycan editing by noncanonical D-amino acids. Microb Drug Resist 18 (3): 306-313.
Cava F, Lam H, de Pedro MA y Waldor MK (2011). Emerging knowledge of regulatory roles of D-amino acids in bacteria. Cell Mol Life Sci 68 (5): 817-831.
Cava F, de Pedro MA, Lam H, Davis BM y Waldor MK (2011). Distinct pathways for modification of the bacterial cell wall by non-canonical D-amino acids. EMBO J 30 (16): 3442-3453.
Lam H, Oh DC, Cava F, Takacs CN, Clardy J, de Pedro MA y Waldor MK (2009). D-amino acids govern stationary phase cell wall remodeling in bacteria. Science 325 (5947): 1552-1555.

