
Foto de grupo. Algunos miembros del grupo GFL de izquierda a derecha y de arriba abajo: Oreto Antúnez, Daniel Medina, Fany Carrasco, Daniele Salmone, Toni Jordán, José García Martínez, Tianlu Li, Alvaro Castells, José E. Pérez Ortín, Ana Miguel, Paula Alepuz, Lucas Morales.
El grupo de Genómica Funcional de Levaduras (GFL, http://www.uv.es/gfl/), establecido inicialmente por el Dr. José E. Pérez Ortín en el Departamento de Bioquímica y Biología Molecular de la Universitat de València (UVEG), comenzó su investigación sobre la biología molecular de levaduras en 1992. A partir de 1995, el trabajo se orientó hacia la genómica de levaduras, participando en proyectos de secuenciación del genoma y de análisis funcional de Saccharomyces cerevisiae. En 2000 se incorporó el Dr. José García Martínez; en 2004, la Dra. Paula Alepuz y más recientemente los Drs. Vicente Tordera y Vicente Arnau. Actualmente, el grupo cuenta también con 3 doctorandos y 1 personal técnico de apoyo a la investigación.
Nuestro grupo ha elegido como organismo modelo para el estudio del control de la expresión génica a la levadura S. cerevisiae, el eucariota mejor conocido. La disponibilidad de técnicas y herramientas genéticas y genómicas hace de este organismo el más flexible para llevar a cabo experimentos sobre las funciones de genes y proteínas para prácticamente todos los procesos fundamentales de las células eucariotas. En la actualidad, llevamos a cabo el estudio del control de la expresión génica en levaduras a través de tres líneas de investigación principales:
Desarrollo de herramientas genómicas para el estudio del recambio de mRNAs
El control de la expresión génica es un proceso de múltiples niveles que afecta a todas las etapas de la transcripción: desde la iniciación a la estabilidad de los mRNAs. La complejidad de este fenómeno requiere, para ser comprendido, nuevas aproximaciones, incluyendo las de escala genómica. Este enfoque solo puede ser realizado en un organismo modelo que tenga las herramientas necesarias, tal como la levadura S. cerevisiae. En nuestro grupo desarrollamos herramientas genómicas en levadura para estudiar la transcripción in vivo y clarificar las redes funcionales que controlan la los genes y la transcripción a escala global. El objetivo de esta línea de investigación es definir los mecanismos que regulan la expresión génica durante la elongación transcripcional y la relación entre los mecanismos de control de la transcripción y la degradación de mensajeros para el control homeostático de sus niveles. Estas herramientas de estudio las estamos adaptando también en la levadura patógena modelo Candida albicans.
Mecanismos de coordinación entre la transcripción y la estabilidad de mRNAs en levaduras durante la respuesta al estrés
Las respuestas al estrés son complejas y abarcan muchos niveles. Uno de los más relevantes es el de la respuesta transcripcional. Con las herramientas genómicas desarrolladas en nuestro laboratorio abordamos la importancia relativa de la síntesis y la degradación de mRNAs en diferentes situaciones de estrés de importancia práctica en biomedicina o biotecnología: la respuesta al estrés osmótico, la respuesta de las cepas vínicas de S. cerevisiae a la deficiencia de nitrógeno en los mostos o la respuesta de C. albicans al estrés oxidativo causado por el hospedador.
Mecanismos moleculares implicados en la regulación post-transcripcional de la expresión génica en respuesta a señales ambientales: regulación del «splicing», la estabilidad y la traducción de mensajeros en respuesta a estrés osmótico en la levadura S. cerevisiae
En los últimos años se ha demostrado que la regulación post-transcripcional ocurre en paralelo a la regulación de la transcripción en respuesta a muchas señales extracelulares y, en muchos casos, especialmente en mamíferos, los mecanismos post-transcripcionales son los que tienen un mayor peso en las respuestas adaptativas. Así, para la regulación de la cantidad de mRNA en la célula se recurre a la variación de la eficiencia de splicing del pre-mRNA y de la tasa de degradación de los mRNAs maduros, lo que sumado a la regulación de sus tasas de traducción permite modificar rápidamente los niveles de proteínas en la célula para generar una respuesta adecuada a cada estímulo. El objetivo de esta línea de investigación es descubrir los mecanismos implicados en la regulación postraduccional de los mRNAs de S. cerevisiae en respuesta a estrés osmótico.
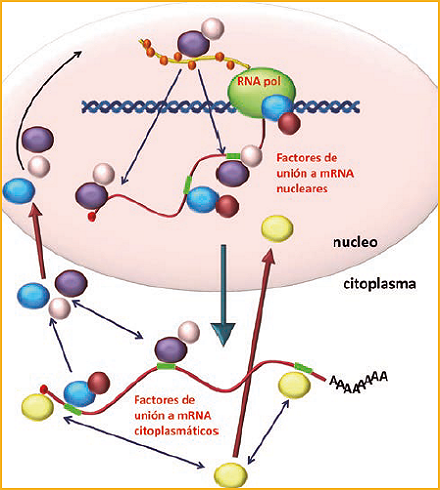
Figura 1. Modelo de intercambio de factores de unión a mRNA durante las distintas etapas del proceso de expresión génica en eucariotas.
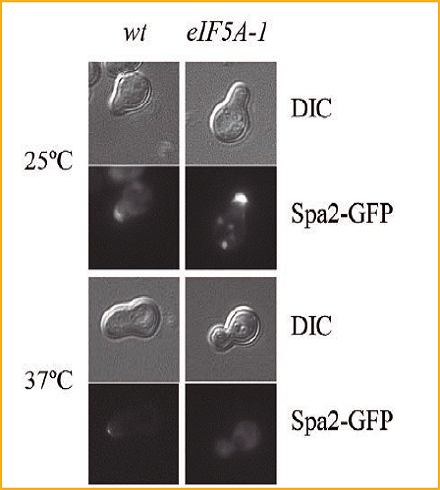
Figura 2. El factor de elongación eIF5A, conservado desde levaduras hasta humanos, es necesario para la formación de shmoos y el crecimiento polarizado en la respuesta a feromona de células de S. cerevisiae. En la foto se muestra cómo la localización en la punta del shmoo de una proteína del polarisoma, Spa2, fusionada a GFP, que se produce tras el tratamiento con feromona, se pierde a temperatura restrictiva en un mutante termosensible de eIF5A.
A lo largo de estos años, hemos establecidos colaboraciones con investigadores de grupos nacionales e internacionales tales como:
- Dr. Joaquín Moreno, Dpto. Bioquímica y Biología Molecular (UVEG).
- Dr. Per Sunnerhagen y Dr. Jonas Warringer. (Göteborg University, Suecia).
- Dr. Gustav Ammerer (University of Vienna, Austria).
- Dr. Sebastián Chávez (Universidad de Sevilla).
- Dr. Francisco Navarro (Universidad de Jaén).
- Dr. Enrique Herrero (Universitat de Lleida).
- Dr. Jesús Pla (Universidad Complutense de Madrid).
- Dr. Joachim Ernst (Heinrich-Heine-Universität Düsseldorf)
- Dr. Joaquín Ariño (Universitat Autònoma de Barcelona)
- Dr. Mordechai Choder (Technion, Israel)
- Dra. Alexandra Mendes-Ferrerira (U. Tras-os-Montes e Alto Douro, Portugal)
En los últimos cinco años hemos recibido financiación a través de cuatro proyectos del Plan Nacional de I+D+i, diez ayudas a la investigación de la Generalitat Valenciana y una subvención de la UVEG. El grupo GFL es miembro de ISIC/ERI BIOTECMED, Instituto Superior de Investigación Cooperativa de la Generalitat Valenciana y Estructura de Investigación Interdisciplinar de la UVEG en Biotecnología y Biomedicina (http://www.uv.es/biotecmed). También forma parte del Microcluster Biotecnología y Biomedicina con Levaduras Modelo (BBLM) compuesto por grupos de la UVEG, la Universitat Politécnica de Valencia, el CSIC y el Centro de Investigación Príncipe Felipe, dentro del Campus de Excelencia Internacional «VLC Campus» (http://www.vlc-campus.com/).
Bibliografía representativa
Magraner-Pardo L, Pelechano V, Coloma MD, Tordera V. (2014). Dynamic remodeling of histone modifications in response to osmotic stress in Saccharomyces cerevisiae. BMC Genomics.15:247.
Li T, Belda-Palazón B, Ferrando A, Alepuz P. (2014). Fertility and Polarized Cell Growth Depends on eIF5A for Translation of Polyproline-Rich Formins in Saccharomyces cerevisiae. Genetics. 197:1191-1200.
Miguel A, Montón F, Li T, Gómez-Herreros F, Chávez S, Alepuz P y Pérez-Ortín JE. (2013). External conditions inversely change the RNA polymerase II elongation rate and density in yeast. Biochim Biophys Acta 11:1248-55.
Nikolaou C, Bermúdez I, Manichanh C, García-Martinez J, Guigó R, Pérez- Ortín JE y Roca J. (2013).
Topoisomerase II regulates yeast genes with singular chromatin architectures. Nucl Acids Res 41: 9243-9256.
Pérez-Ortín JE, Alepuz P, Chávez S y Choder M. (2013). Eukaryotic mRNA decay: methodologies, pathways, and links to other stages of gene expression. J Mol Biol. 425:3750-3775
Pérez-Ortín JE, Medina DA, Chávez S y Moreno J. (2013). What do you mean by transcription rate?: The conceptual difference between nascent transcription rate and mRNA synthesis rate is essential for the proper understanding of transcriptomic analyses. Bioessays 35:1056-1062.
Haimovich G, Medina DA, Causse SZ, Garber M, Millán-Zambrano G, Barkai O, Chávez S, Pérez-Ortín JE, Darzacq X y Choder M. (2013). Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. Cell 153:1000-1011.
Garre E, Romero-Santacreu L, Barneo-Muñoz M, Miguel A, Pérez-Ortín JE y Alepuz P. (2013). Nonsense-mediated mRNA decay controls the changes in yeast ribosomal protein pre-mRNAs levels upon osmotic stress. PLoS ONE DOI: 10.1371/journal.pone.0061240.
Alic A, Pérez-Ortín JE, Moreno J y Arnau V. (2013). mRNAStab–a web application for mRNA stability analysis. Bioinformatics 29:813-814.
García-Martínez, J, Ayala, G, Pelechano, V, Chávez, S, Herrero, E y Pérez-Ortín, JE. (2012). The relative importance of transcription rate, cryptic transcription and mRNA stability on shaping stress responses in yeast. Transcription 3: 39-44.
Pérez-Ortín, JE, De Miguel-Jiménez y Chávez, S (2012). Genome-wide studies of mRNA synthesis and degradation in eukaryotes. Biochim. Biophys. Acta 1819: 604-615.
Castells-Roca L, García-Martínez J, Moreno J, Herrero E, Bellí, G y Pérez-Ortín, JE. (2011). Heat shock response in yeast involves changes in both transcription rates and mRNA stabilities. PLoS ONE 6: e17272.
García-López, MC, Pelechano V, Mirón-García MC. Garrido-Godino AI, Werner M, Pérez-Ortín JE y Navarro F. (2011). The Conserved Foot Domain of RNA Pol II Associates with Proteins Involved in Transcriptional Initiation and/or Early Elongation. Genetics 189: 1235-1248.
García-Martínez J, Ayala G, Pelechano V, Chávez S, Herrero E y Pérez-Ortín JE. (2012). The relative importance of transcription rate, cryptic transcription and mRNA stability on shaping stress responses in yeast. Transcription 3: 39-44.
Pérez-Ortín JE., De Miguel-Jiménez y Chávez S. (2012). Genome-wide studies of mRNA synthesis and degradation in eukaryotes. Biochim. Biophys. Acta 1819: 604-615.
Rodríguez-Gil A, García-Martínez J, Pelechano V, Muñoz-Centeno M.C, Geli V, Pérez-Ortín JE. y Chávez S. (2010). The distribution of active RNA polymerase II along the transcribed region is gene-specific and controlled by elongation factors. Nucleic Acids Res. 38:4651-64
Pelechano V, Chávez S. y Pérez-Ortín JE. (2010). A complete set of nascent transcription rates for yeast genes. PLoS ONE 5: e15442.
Romero-Santacreu L, Orozco H, Garre E y Alepuz P. (2010). The bidirectional cytomegalovirus immediate/early promoter is regulated by Hog1 and the stress transcription factors Sko1 and Hot1 in yeast. Mol. Gen. Genom. 283: 510-518.
Romero-Santacreu L, Moreno J, Pérez-Ortín,JE y Alepuz P. (2009). Specific and global regulation of mRNA stability during osmotic stress in Saccharomyces cerevisiae. RNA 15:1110–1120.
Pelechano V, Jimeno-González S, Rodríguez-Gil A, García-Martínez J, Pérez-Ortín, JE y Chávez S. (2009). Regulon-specific control of transcription elongation across the yeast genome. PLoS Genet. 5(8):e1000614.
