https://www.ucm.es/signalyeast/

Foto de grupo. De izquierda a derecha, Óscar Barbero-Úriz, Humberto Martín, María Molina, Víctor J. Cid, Graciela Alonso, Alejandro Fernández-Vega, Beatriz Lavilla-García, Teresa Fernández-Acero, Isabel Rodríguez-Escudero y Sara López-Montesino
Nuestro laboratorio se ubica en el corazón del campus de Moncloa de la Universidad Complutense. En torno al eje común de las rutas de señalización celular en eucariotas, trabajamos en distintas líneas, unas con una orientación básica centradas en el estudio de rutas de señalización en la levadura Saccharomyces cerevisiae y otras aplicadas al desarrollo de este modelo celular para estudios genéticos y moleculares de patologías humanas.
Las rutas de señalización son esenciales para todas las células, desde los sistemas procariotas más elementales hasta los eucariotas superiores más complejos. Permiten la detección y adaptación a cambios fisicoquímicos en el medio ambiente, así como la comunicación intercelular. Altamente conservadas a lo largo de la evolución, estas vías responden a diversos estímulos para regular procesos fisiológicos cruciales para la supervivencia celular y el control de la proliferación. Desde la clonación y caracterización hace tres décadas del gen SLT2, que codifica la MAPK (proteína quinasa activada por mitógenos) responsable del mantenimiento de la integridad de la pared celular (CWI), la línea de investigación dirigida por Teresa Fernández-Acero, Humberto Martín y María Molina, se ha enfocado al estudio de la función de diversos componentes de esta vía, los mecanismos que controlan su activación y regulación, especialmente los relacionados con la fosforilación/desfosforilación, así como su interacción con otras vías de señalización.
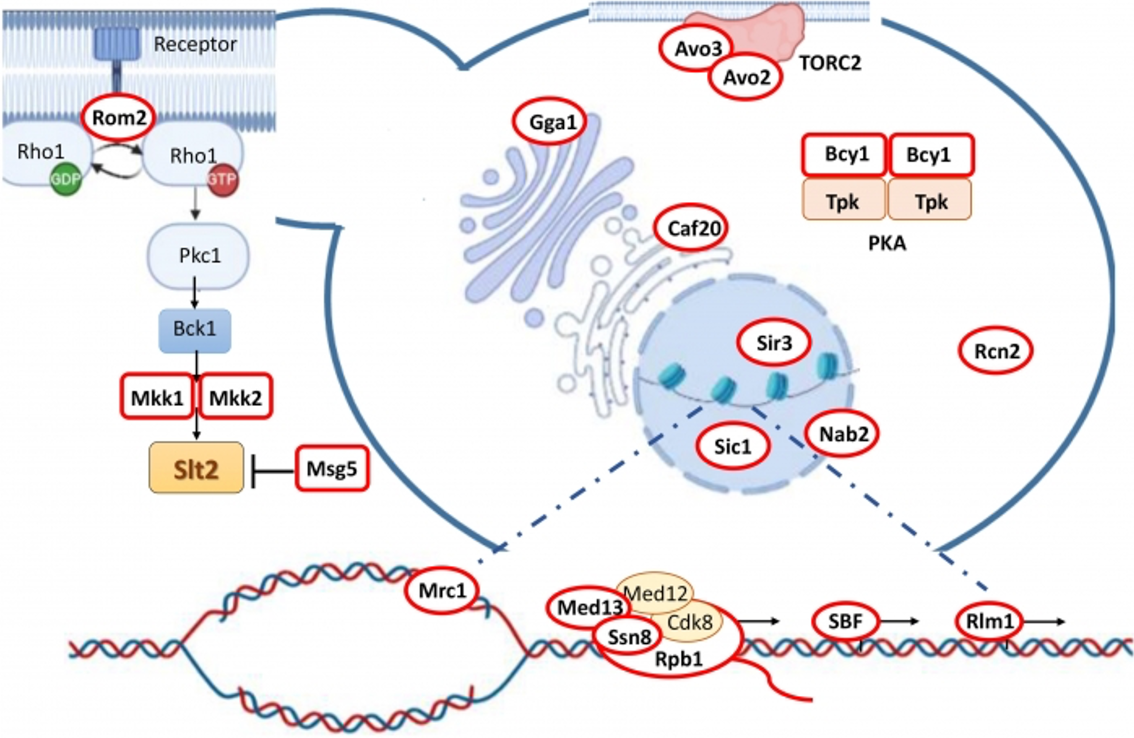
Figura 1. Esquema de la ruta CWI y de los sustratos de Slt2 (rodeados en rojo) en diferentes localizaciones celulares (González-Rubio et al. 2022. J Fungi 8:368).
Por otra parte, el conocimiento sobre la señalización celular en la homeostasis de la levadura nos permite reprogramar el sistema mediante la introducción de componentes heterólogos. En este contexto el grupo, en una línea dirigida por los Dres. Isabel Rodríguez Escudero, Víctor J. Cid y María Molina, trabajamos en la elucidación de la función de proteínas de virulencia de microorganismos patógenos intracelulares mediante su expresión en S. cerevisiae y su interacción con funciones procarióticas, con énfasis en la señalización celular. A lo largo de la historia del grupo hemos colaborado con otros laboratorios en el estudio de efectores de Legionella, Klebsiella, Brucella, Coxiella, Salmonella y Escherichia coli enteropatogénica implicados en el establecimiento del nicho intracelular y la evasión del sistema inmunitario.
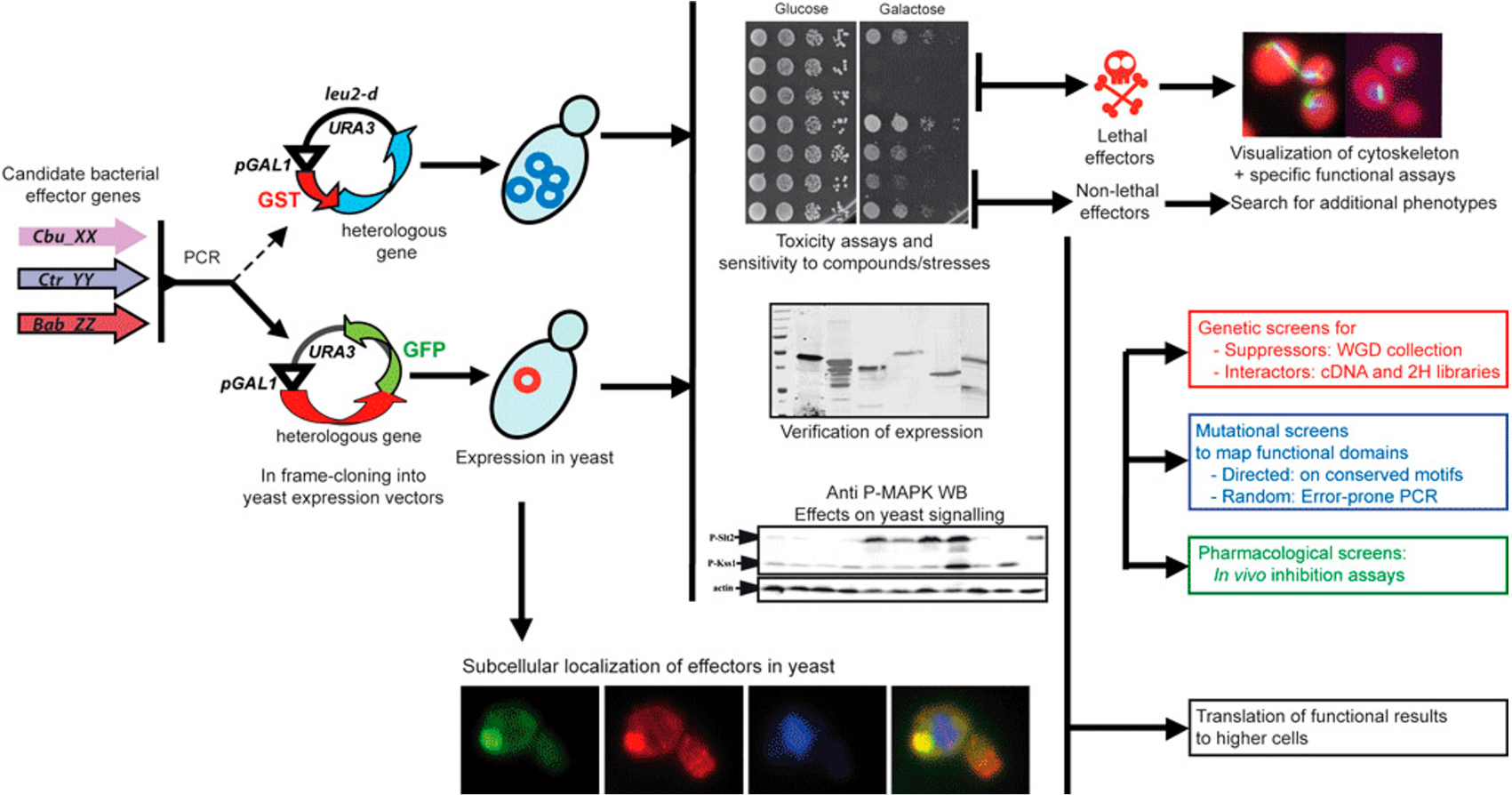
Figura 2. Diagrama de trabajo para estudios en levadura de efectores de virulencia bacterianos de función desconocida.
Otra línea aplicada del laboratorio, dirigida por los Dres. Teresa Fernández-Acero, Víctor J. Cid y María Molina se centra en aplicar estrategias de biología sintética para reconstituir rutas de señalización celular humanas en el chasis de la levadura. En este contexto hemos reconstituido en este modelo la vía oncogénica humana PI3K, lo que ha permitido el desarrollo de ensayos funcionales sobre dicho oncogen, su efector, la proteína quinasa Akt y el supresor de tumores PTEN. Actualmente, en colaboración con investigadores de Harvard Medical School y otros laboratorios, estamos trabajando en el ensamblaje de signalosomas humanos relacionados con la inmunidad innata, como el myddosoma en la señalización de TLR, el inflamasoma o la vía cGAS-STING.
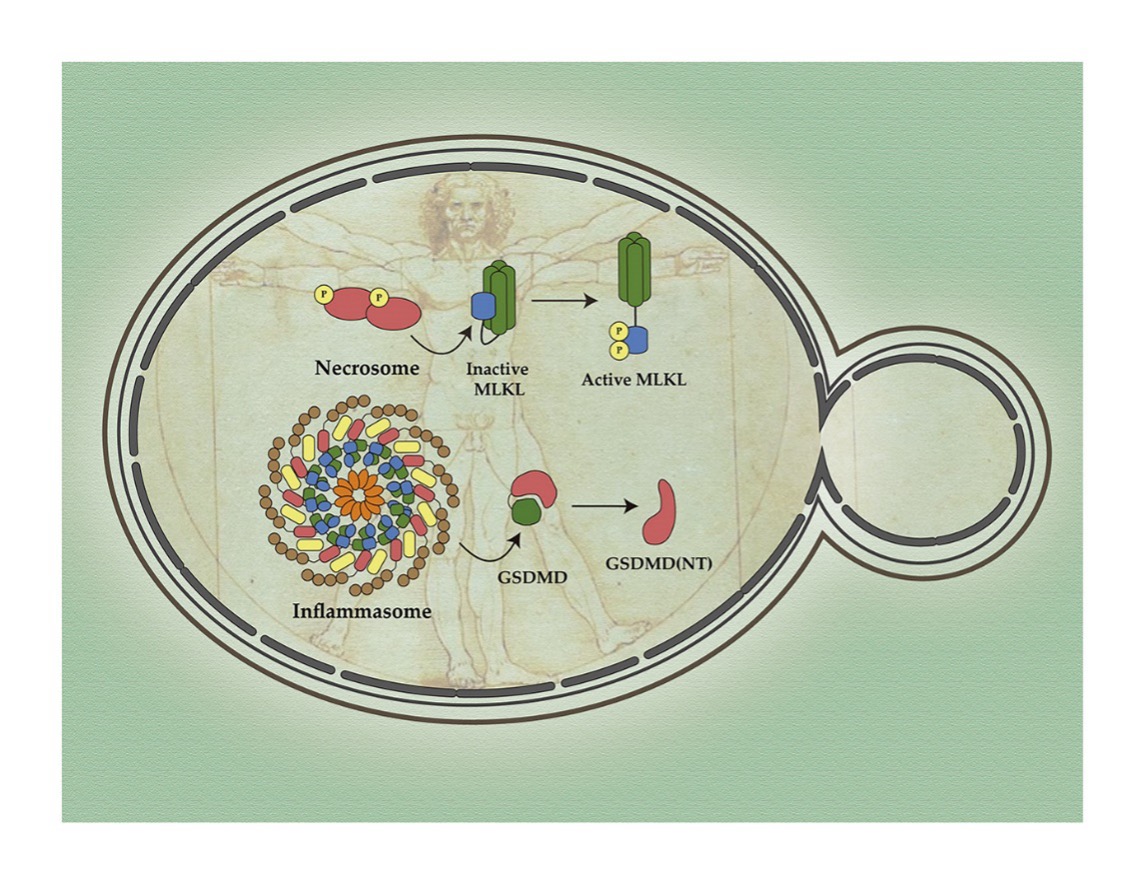
Figura 3. El ensamblaje de rutas de inmunidad innata y muerte celular regulada en el modelo de levadura mediante expresión heteróloga de sus componentes permite el desarrollo de plataformas para estudios genéticos y farmacológicos (Barbero-Úriz et al. 2025. Biomolecules. 15:530)
Principales artículos del grupo en los últimos 5 años:
Barbero-Úriz Ó, Valenti M, Molina M, Fernández-Acero T, Cid VJ. (2025). Modeling Necroptotic and Pyroptotic Signaling in Saccharomyces cerevisiae. Biomolecules. 15(4):530. doi: 10.3390/biom15040530.
Valenti M, Molina M, Cid VJ. (2023). Human gasdermin D and MLKL disrupt mitochondria, endocytic traffic and TORC1 signalling in budding yeast. Open Biol.13: 220366. doi.org/10.1098/rsob.220366
González-Rubio G, Martín H, Molina M. (2023) The Mitogen-Activated Protein Kinase Slt2 Promotes Asymmetric Cell Cycle Arrest and Reduces TORC1-Sch9 Signaling in Yeast Lacking the Protein Phosphatase Ptc1. Microbiol Spectr. e0524922. doi: 10.1128/spectrum.05249-22.
Sá-Pessoa J, López-Montesino S, Przybyszewska K, Rodríguez-Escudero I, Marshall H, Ova A, Schroeder GN, Barabas P, Molina M, Curtis T, Cid VJ, Bengoechea JA. (2023). A trans-kingdom T6SS effector induces the fragmentation of the mitochondrial network and activates innate immune receptor NLRX1 to promote infection. Nat Commun. 14(1):871. doi: 10.1038/s41467-023-36629-3. PMID: 36797302.
Torices L, Mingo J, Rodríguez-Escudero I, Fernández-Acero T, Luna S, Nunes-Xavier CE, López JI, Mercadillo F, Currás M, Urioste M, Molina M, Cid VJ, Pulido R. (2022) Functional analysis of PTEN variants of unknown significance from PHTS patients unveils complex patterns of PTEN biological activity in disease. Eur J Hum Genet. doi: 10.1038/s41431-022-01265-w.
de Oya IG, Jiménez-Gutiérrez E, Gaillard H, Molina M, Martín H, Wellinger RE. (2022). Manganese Stress Tolerance Depends on Yap1 and Stress-Activated MAP Kinases. Int J Mol Sci. 23:15706. doi: 10.3390/ijms232415706.
Jiménez-Gutiérrez E, Fernández-Acero T, Alonso-Rodríguez E, Molina M, Martín H. (2022) Neomycin Interferes with Phosphatidylinositol-4,5-Bisphosphate at the Yeast Plasma Membrane and Activates the Cell Wall Integrity Pathway. Int J Mol Sci. ;23(19):11034. doi: 10.3390/ijms231911034.
González-Rubio G, Sastre-Vergara L, Molina M, Martín H, Fernández-Acero T. (2022) Substrates of the MAPK Slt2: Shaping Yeast Cell Integrity. J Fungi (Basel). 8(4):368. doi: 10.3390/jof8040368.
Sellers-Moya Á, Nuévalos M, Molina M, Martín H. (2021). Clotrimazole-Induced Oxidative Stress Triggers Novel Yeast Pkc1-Independent Cell Wall Integrity MAPK Pathway Circuitry. J Fungi (Basel). 7(8):647. doi: 10.3390/jof7080647.
Coronas-Serna, JM, del Val E, Kagan JC, Molina M, Cid VJ (2021). Heterologous Expression and Assembly of Human TLR Signaling Components in Saccharomyces cerevisiae. Biomolecules, 11(11), 1737; doi.org/10.3390/biom11111737
Valenti M, Molina M, Cid VJ. (2021). Heterologous Expression and Auto-Activation of Human Pro-Inflammatory Caspase-1 in Saccharomyces cerevisiae and Comparison to Caspase-8. Front Immunol. 12:668602 doi: 10.3389/fimmu.2021.668602.
Luna S, Torices L, Mingo J, Amo L, Rodríguez-Escudero I, Ruiz-Ibarlucea P, Erramuzpe A, Cortés JM, Tejada MI, Molina M, Nunes-Xavier CE, López JI, Cid VJ, Pulido R. (2021). A global analysis of the reconstitution of PTEN function by translational readthrough of PTEN pathogenic premature termination codons. Hum Mutat. 42(5):551-566. doi: 10.1002/humu.24186.
González-Rubio G, Sellers-Moya Á, Martín H, Molina M. (2021). A walk-through MAPK structure and functionality with the 30-year-old yeast MAPK Slt2. Int Microbiol. doi: 10.1007/s10123-021-00183-z.
González-Rubio G, Sellers-Moya Á, Martín H, Molina M. (2021) Differential Role of Threonine and Tyrosine Phosphorylation in the Activation and Activity of the Yeast MAPK Slt2. Int J Mol Sci. 23;22(3):1110. doi: 10.3390/ijms22031110.PMID: 33498635
Coronas-Serna JM, Louche A, Rodríguez-Escudero M, Roussin M, Imbert PRC, Rodríguez-Escudero I, Terradot L,Molina M, Gorvel JP, Cid VJ, Salcedo SP. (2020) The TIR-domain containing effectors BtpA and BtpB from Brucella abortus impact NAD metabolism. PLoS Pathogens. 16(4):e1007979. doi: 10.1371/journal.ppat.1007979. PMID: 32298382.
Storey D, McNally A, Åstrand M, Sa-Pessoa Graca Santos J, Rodriguez-Escudero I, Elmore B, Palacios L, Marshall H, Hobley L,Molina M, Cid VJ, Salminen TA, Bengoechea JA. (2020) Klebsiella pneumoniae type VI secretion system-mediated microbial competition is PhoPQ controlled and reactive oxygen species dependent. PLoS Pathog. 16(3):e1007969. doi: 10.1371/journal.ppat.1007969. PMID: 32191774.
Jiménez-Gutiérrez E, Alegría-Carrasco E, Alonso-Rodríguez E, Fernández-Acero T, Molina M, Martín H. (2020). Rewiring the yeast cell wall integrity (CWI) pathway through a synthetic positive feedback circuit unveils a novel role for the MAPKKK Ssk2 in CWI pathway activation. FEBS J. doi: 10.1111/febs.15288. PMID: 32150787.
