
Foto de grupo. De izquierda a derecha: Antonio Ventosa, Maribel Reina, Blanca Vera, Cristina Sánchez-Porro, Ana Durán, Rafael R. de la Haba, Cristina Galisteo
Nuestro grupo de investigación ha venido realizando estudios metagenómicos en ambientes hipersalinos, especialmente en salinas costeras de nuestro país, que constituyen excelentes modelos de estudio de hábitats extremos, presentando un gradiente de salinidad desde la del agua de mar hasta la saturación de sales. Estos estudios han permitido determinar la estructura y actividad de los grupos de arqueas y bacterias que constituyen la diversidad microbiana de estos sistemas hipersalinos, de los cuales un porcentaje elevado de microorganismos abundantes en estos ambientes aún no han sido aislados en cultivo puro. Con tal motivo, un objetivo de nuestro grupo consiste en realizar estudios exhaustivos enfocados al aislamiento y caracterización de dichos grupos de arqueas y bacterias halófilas a partir de salinas marinas.
Entre los nuevos grupos que hemos aislado y caracterizado en detalle recientemente se encuentran especies de los géneros Natronomonas, Halorientalis, Natrinema y Halorubrum, y muy especialmente debemos referirnos al aislamiento de tres nuevas cepas pertenecientes al género Halonotius y cuatro nuevas cepas que constituyen un nuevo género, para el que proponemos la designación de Halosegnis, con dos nuevas especies: Halosegnis longus y Halosegnis rubeus. Estudios de reclutamiento genómico y de comparación de secuencias ribosómicas indican que estos nuevos taxones constituyen grupos abundantes en ambientes hipersalinos, con una amplia distribución geográfica. Las reconstrucciones metabólicas basadas en el análisis de los genomas de las especies del género Halonotius (Figura 1) demostró que poseen los genes de biosíntesis de cobalamina (vitamina B12), un resultado relevante que permitiría justificar su amplia distribución geográfica y abundancia en ambientes hipersalinos, en los cuales podrían desempeñar un importante papel como fuente de dicha vitamina. Estos estudios forman parte de la Tesis Doctoral de Ana Durán Viseras, que será presentada en fechas recientes.
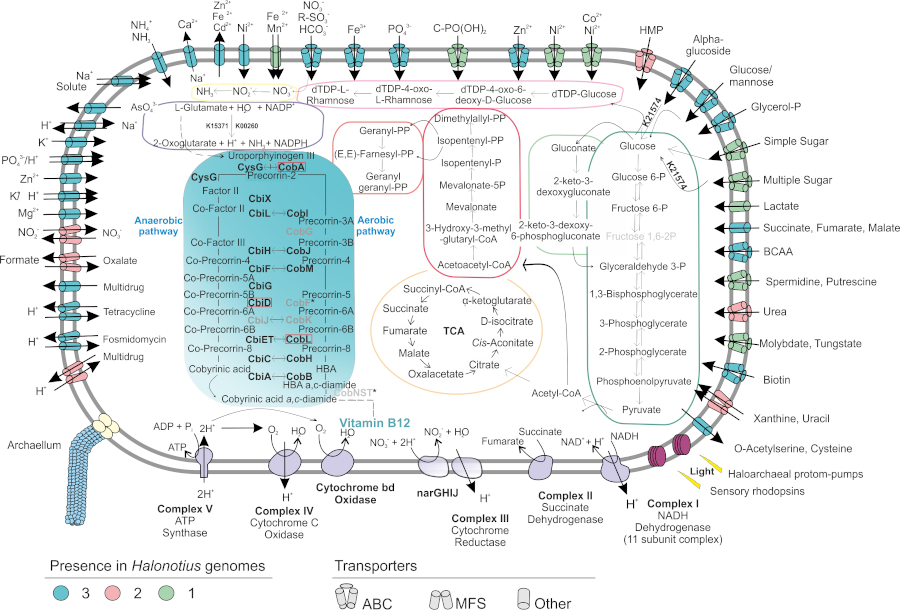
Figura 1. Reconstrucción metabólica de haloarqueas del género Halonotius (tomada de Durán-Viseras et al., Front. Microbiol. 10: 1928, 2019).
Diversidad filogenómica y funcional de procariotas en suelos hipersalinos
Un proyecto de investigación que actualmente estamos desarrollando tiene como objetivos la determinación de la estructura y actividad de la comunidad procariota de suelos hipersalinos, así como la comparación de dichos ambientes terrestres con los ambientes hipersalinos acuáticos que hemos estudiado previamente y de los que existe mucha mayor información. Este estudio ha sido objeto de una Tesis Doctoral, realizada por Blanca Vera Gargallo (2018), en suelos hipersalinos localizados en las Marismas del Odiel, Huelva, mediante el empleo de técnicas metagenómicas, metagenéticas y de marcaje con isótopos estables (SIP). Los suelos hipersalinos estudiados mostraron una mayor diversidad tanto taxonómica como funcional con respecto a los ambientes acuáticos estudiados con anterioridad, siendo mayoritarios grupos pertenecientes a Euryarchaeota, Bacteroidetes, Balneolaeota y Rhodothermaeota, si bien una elevada proporción de secuencias metagenómicas se afiliaban a taxones no cultivados hasta la fecha, especialmente pertenecientes al dominio Bacteria. Los estudios de SIP indican que la comunidad de arqueas de los suelos salinos estudiados se encuentra mejor adaptada que la comunidad bacteriana presente en dichos ambientes extremos (Vera-Gargallo & Ventosa, 2018; Vera-Gargallo et al., 2019).
Género Salinivibrio: MLSA y genómica comparativa
El género Salinivibrio incluye bacterias halófilas moderadas, que se encuentran normalmente en ambientes hipersalinos (salinas, lagos salinos), así como en salmueras y alimentos en salazón. Este género incluye un número muy reducido de especies y nuestro grupo de investigación ha venido realizando estudios acerca de la situación taxonómica de las mismas, así como el aislamiento de nuevas cepas de Salinivibrio que pudieran constituir nuevas especies de dicho género. Con dicha finalidad, y como parte del estudio de Tesis Doctoral de Clara López Hermoso (2017), aislamos 70 nuevas cepas de ambientes salinos de diferentes localizaciones. Debido a la dificultad para diferenciar dichas cepas a nivel de especie mediante la comparación de secuencias del ARNr 16S, establecimos una metodología alternativa, basada en un análisis por secuenciación multilócica (MultiLocus Sequence Analysis, MLSA), mediante la comparación de los genes “housekeeping” gyrB, recA, rpoA y rpoB. Este estudio fue validado mediante la realización de estudios alternativos basados en las técnicas convencionales de hibridación ADN-ADN, usadas tradicionalmente en la delineación de especies en procariotas (López-Hermoso et al., 2017). Más recientemente, hemos obtenido los “draft” genomas de 36 cepas de Salinivibrio, incluyendo las cepas tipo de todas las especies y subespecies descritas. Por otro lado, al no disponer de ningún genoma cerrado de representantes de Salinibibrio, hemos procedido a la secuenciación del genoma completo de la cepa tipo de Salinivibrio kushneri, una especie descrita recientemente por nuestro grupo de investigación. Al igual que otros miembros de la familia Vibrionaceae, S. kushneri posee dos cromosomas circulares con tamaños de 2,84 Mb y 0,60 Mb, respectivamente; por otro lado, posee un elevado número (9) de operones ribosómicos, típico de bacterias con tasas de crecimiento rápido en medios de laboratorio. El análisis de los genomas de las cepas de Salinivibrio puso de manifiesto una alta homogeneidad y grado de sintenia entre los representantes de este género. El estudio filogenómico ha permitido conocer con detalle la estructura del género, quedando constituido por seis especies (S. costicola, S. siamensis, S. sharmensis, S. proteolyticus, S. kushneri y S. socompensis) y una especie adicional, que debe ser descrita en el futuro (de la Haba et al., 2019).
Publicaciones seleccionadas (últimos 5 años)
Ventosa A, de la Haba RR, Sánchez-Porro, C, Papke, RT (2015). Microbial diversity of hypersaline environments: a metagenomic approach. Curr. Op. Microbiol. 25: 80-87.
Amoozegar MA, Siroosi M, Atashgahi S, Smidt H, Ventosa A. (2017). Systematics of haloarchaea and biotechnological potential of their hydrolytic enzymes. Microbiology 163: 623-645.
López-Hermoso C, de la Haba RR, Sánchez-Porro C, Papke RT, Ventosa A (2017). Assessment of Multilocus Sequence Analysis as a valuable tool for the classification of the genus Salinivibrio. Front. Microbiol. 8: 1107.
Oren A, Ventosa A, Kamekura M (2017). Class Halobacteria. Bergey’s Manual of Systematics of Archaea and Bacteria. W. B. Whitman (ed.), pp. 1-5. Whiley & Sons, New Jersey.
León MJ, Hoffmann T, Sánchez-Porro C, Heider J, Ventosa A, Bremer E (2018). Compatible solute synthesis and import by the moderate halophile Spiribacter salinus: physiology and genomics. Front. Microbiol. 9: 108.
Vera-Gargallo B, Ventosa, A (2018). Metagenomic insights into the phylogenetic and metabolic diversity of the prokaryotic community dwelling in hypersaline soils from the Odiel saltmarshes (SW Spain). Genes 9: 152.
de la Haba RR, Corral P, Sánchez-Porro C, Infante-Domínguez C, Makkay A, Amoozegar MA, Ventosa A, Papke RT (2018). Genotypic and lipid analyses of strains from the archaeal genus Halorubrum reveal insights into their taxonomy, divergence and population structure. Front. Microbiol. 9: 512.
Vera-Gargallo B, Chowdhury TR, Brown J, Fansler SJ, Durán-Viseras A, Sánchez-Porro C, Bailey VL, Jansson JK, Ventosa A (2019). Spatial distribution of prokaryotic communities in hypersaline soils. Sci. Rep. 9: 1769.
Ventosa A, de la Haba RR, Sánchez-Porro C (2019). Genus Haloarcula. Bergey’s Manual of Systematics of Archaea and Bacteria. W. B. Whitman (ed.), pp. 1-12. Whiley & Sons, New Jersey.
Amoozegar MA, Safarpour A, Noghabi KA, Bakhtiary T, Ventosa A. (2019). Halophiles and their vast potential in biofuel production. Front. Microbiol. 10: 1895.
Durán-Viseras A, Andrei A-S, Ghai R, Sánchez-Porro C, Ventosa A (2019). New Halonotius species provide genomics-based insights into cobalamin synthesis in haloarchaea. Front. Microbiol. 10: 1928.
de la Haba RR, López-Hermoso C, Sánchez Porro C, Konstantinidis K, Ventosa A (2019). Comparative genomics and phylogenomic analysis of the genus Salinivibrio. Front. Microbiol. 10: 2104.
