
Foto de grupo. De izquierda a derecha: Sara Belén Hernández Piñero, Agustín Vioque Peña, Alicia M. Muro Pastor, Isidro Álvarez Escribano y Belén Suárez Murillo.
Las cianobacterias son un grupo de microorganismos fotosintéticos con requerimientos nutricionales muy reducidos y una enorme capacidad de adaptación a entornos cambiantes, por lo que son objeto de interés biotecnológico para la producción sostenible de distintos compuestos, incluyendo biocombustibles. Los mecanismos de adaptación de las cianobacterias a la deficiencia de nitrógeno están controlados por el regulador transcripcional NtcA, que regula el uso de distintas fuentes de nitrógeno, incluyendo en última instancia la posibilidad de fijar nitrógeno atmosférico (N2) mediante la nitrogenasa, una enzima extremadamente sensible al oxígeno cuya actividad es incompatible con la fotosintesis oxigénica. En cianobacterias complejas, como nuestro organismo modelo Nostoc sp. PCC 7120, esta incompatibilidad se resuelve confinando la nitrogenasa en unas células especializadas denominadas heterocistos. En condiciones de fijación de nitrógeno los filamentos se comportan como organismos pluricelulares con división de tareas entre dos tipos celulares interdependientes, las células vegetativas y los heterocistos (figura 1) (Muro-Pastor & Hess 2012). La diferenciación de heterocistos, que está en última instancia controlada por NtcA, depende también de HetR, un regulador específico de diferenciación celular. Nuestro trabajo se centra en el estudio de la participación de RNAs reguladores, tanto pequeños RNAs (sRNAs) como RNAs antisentido en los procesos de adaptación al estrés de carencia de nitrógeno y la diferenciación de heterocistos.
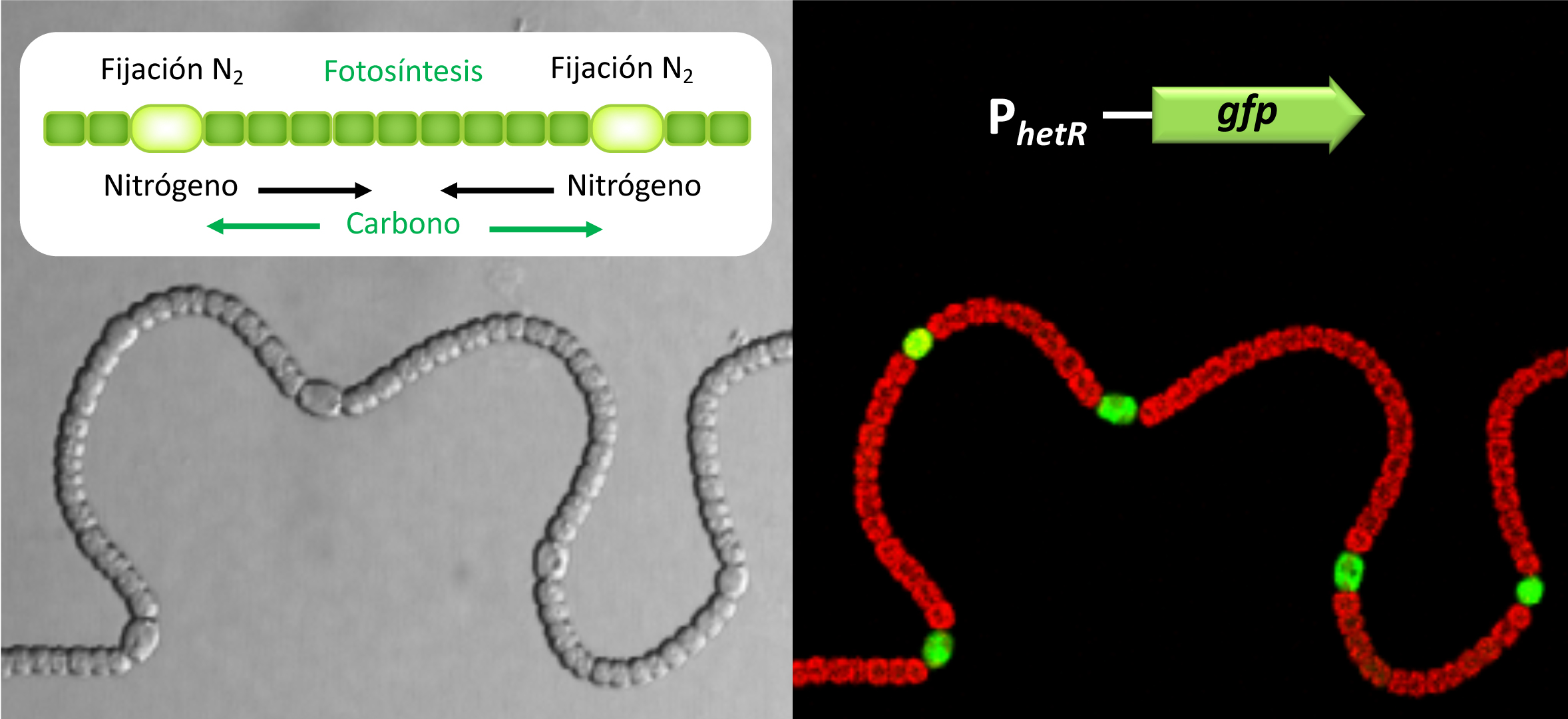
Figura 1. Las cianobacterias formadoras de heterocistos son organismos multicelulares con división de tareas metabólicas. Se muestran las relaciones funcionales entre heterocistos (células especializadas en la fijación de nitrógeno atmosférico) y células vegetativas (que llevan a cabo la fijación fotosintética del CO2). La expresión de la proteína testigo GFP desde el promotor del regulador de la diferenciación HetR ilustra la existencia de patrones transcripcionales exclusivos de heterocistos. La fluorescencia roja corresponde a los pigmentos fotosintéticos de las células vegetativas.
Hemos definido el “paisaje transcripcional” de nuestro organismo modelo mediante una metodología de RNASeq que evidenció abundante transcripción no codificante. La inclusión de una estirpe mutante hetR, que no es capaz de diferenciar heterocistos, nos permitió clasificar las respuestas transcripcionales a la deficiencia de nitrógeno en dos grandes regulones, el regulón NtcA, de respuestas globales, y el regulón HetR, que incluye respuestas específicamente relacionadas con la diferenciación de heterocistos (Mitschke et al. 2011). En ambos regulones hemos identificado moléculas de RNA potencialmente reguladoras.
Utilizando la información derivada del análisis transcriptómico hemos diseñado un acercamiento informático para la predicción de secuencias transcritas como sRNAs a partir de regiones intergénicas (Brenes-Álvarez et al. 2016). Este acercamiento implementa un análisis de conservación filogenética que sugiere que algunos de los sRNAs predichos se transcribirían de hecho como RNAs funcionales. Así, por ejemplo, NsrR1 (nitrogen stress-repressed RNA 1) es un sRNA de 45 nucleótidos, codificado en todos los genomas de estirpes formadoras de heterocistos, cuya expresión es reprimida por NtcA y que participa en la regulación de la expresión de la proteína NblA, implicada en el reciclaje de aminoácidos en condiciones de carencia de nitrógeno (Álvarez-Escribano et al. 2018). De esta forma, la regulación de los niveles de la proteína NblA estaría operada por un circuito mixto en el que participan un factor transcripcional (NtcA) y un sRNA (NsrR1). También hemos analizado NsiR4 (nitrogen stress-induced RNA4), que se transcribe desde un promotor activado por NtcA y que en una cianobacteria unicelular participa en la regulación de la asimilación de amonio a través de la modulación de la traducción de la proteína IF7, un factor inactivante de la glutamina sintetasa (Klähn et al. 2015). La versión de NsiR4 presente en cianobacterias complejas formadoras de heterocistos modula actividades enzimáticas implicadas en la asimilación de CO2 (Brenes-Álvarez et al., 2021).
Hemos diseñado también un acercamiento global a la identificación de transcritos específicos de los heterocistos, entre los que hemos encontrado tanto sRNAs como RNAs antisentido que podrían ejercer un papel regulador. A partir de datos de hibridación de microarrays hemos construido una red de co-expresión y llevado a cabo un análisis de “clustering” en el que los diferentes transcritos aparecen agrupados en función de patrones transcripcionales comunes (Brenes-Álvarez et al. 2019). Entre los transcritos transcripcionalmente agrupados con los de genes implicados en diferenciación de heterocistos aparece por ejemplo NsiR1 (nitrogen stress-induced RNA1), un sRNA de 60 nucleótidos que se puede considerar un marcador muy temprano de células en diferenciación (Muro-Pastor 2014) que hemos demostrado que afecta las etapas iniciales de la diferenciación de heterocistos (Brenes-Álvarez et al., 2020, 2022). Asimismo, entre los transcritos que se expresan exclusivamente en heterocistos aparecen varios transcritos antisentido, como el as_glpX, que se transcribe en la hebra contraria del gen que codifica la sedoheptulosa-1,7-bisfosfatasa, una enzima requerida para la fijación fotosintética de CO2 o el as_gltA, que se transcribe en la hebra contraria del gen que codifica la citrato sintasa. La transcripción de estos RNAs antisentido específicamente en los heterocistos contribuiría a la reprogramación metabólica que acompaña la diferenciación de estas células especializadas (Olmedo-Verd et al. 2019, Brenes-Álvarez et al., 2023).
En resumen, en los últimos años hemos contribuido a definir circuitos reguladores mixtos que implican a los reguladores transcripcionales NtcA y HetR y a moléculas de RNA no codificantes (tanto pequeños RNAs como RNAs antisentido) cuya expresión está bajo control de los mismos. Nuestro trabajo se lleva a cabo en colaboración con el laboratorio dirigido por Wolfgang R. Hess (Genetics and Experimental Bioinformatics, Universidad de Freiburg, Alemania) y está financiado actualmente por la Agencia Estatal de Investigación en el marco del Programa Estatal de Generación de Conocimiento y Fortalecimiento Científico y Tecnológico del Sistema de I+D+i (PID2022-138128NB-I00).
https://www.ibvf.us-csic.es/grupos-investigacion/rnas-reguladores-de-cianobacterias/
Bibliografía representativa
Álvarez-Escribano I, Suárez-Murillo B, Brenes-Álvarez M, Vioque A, Muro-Pastor AM. (2024) Antisense RNA regulates glutamine synthetase in a heterocyst-forming cyanobacterium. Plant Physiol. 195:2911-2920.
Brenes-Álvarez, M., Vioque, A., Muro-Pastor, A. M. (2023) Nitrogen-regulated antisense transcription in the adaptation to nitrogen deficiency in Nostoc. PNAS Nexus, 2: pgad187.
Brenes-Álvarez, M., Vioque, A., Muro-Pastor, A. M. (2022) The heterocyst-specific small RNA NsiR1 regulates the commitment to differentiation in Nostoc. Microbiology Spectrum 10: e02274-21.
Song, K., Baumgartner, D., Hagemann, M., Muro-Pastor, A. M., Maaß, S., Becher, D., and Hess W. R. (2022) AtpΘ is an inhibitor of F0F1 ATP synthase to arrest ATP hydrolysis during low-energy conditions in cyanobacteria. Current Biology 32: 136-148.
Brenes-Álvarez M, Olmedo-Verd E, Vioque A, Muro-Pastor AM. (2021) A nitrogen stress inducible small RNA regulates CO2 fixation in Nostoc. Plant Physiology, 187:787-798.
Álvarez-Escribano I, Brenes-Álvarez M, Olmedo-Verd E, Georg J, Hess WR, Vioque A, Muro-Pastor, AM (2021) NsiR3, a nitrogen stress-inducible small RNA, regulates proline oxidase expression in the cyanobacterium Nostoc sp. PCC 7120. The FEBS Journal 288: 1614-1629.
Brenes-Álvarez M, Minguet M, Vioque A, Muro-Pastor AM. (2020) NsiR1, a small RNA with multiple copies, modulates heterocyst differentiation in the cyanobacterium Nostoc sp. PCC 7120. Environmental Microbiology 22: 3325-3338.
Brenes-Álvarez M, Vioque A, Muro-Pastor AM. (2020) The integrity of the cell wall and its remodeling during heterocyst differentiation is regulated by phylogenetically conserved sRNA Yfr1 in Nostoc sp. PCC 7120. mBio 11: e02599-19.
Olmedo-Verd E, Brenes-Álvarez M, Vioque A, Muro-Pastor AM. (2019). A heterocyst-specific antisense RNA contributes to metabolic reprogramming in Nostoc sp. PCC 7120. Plant Cell Physiol 60: 1646-1655.
Brenes-Álvarez M, Mitschke J, Olmedo-Verd E, Georg J, Hess WR, Vioque A, Muro-Pastor AM. (2019). Elements of the heterocyst-specific transcriptome unravelled by co-expression analysis in Nostoc sp. PCC7120. Environ Microbiol 21: 2544-2558.
Álvarez-Escribano I, Vioque A, Muro-Pastor AM. (2018). NsiR1, a nitrogen stress-repressed sRNA, contributes to the regulation of nblA in Nostoc sp. PCC 7120. Front Microbiol 9: 2267.
Brenes-Álvarez M, Olmedo-Verd E, Vioque A, Muro-Pastor AM. (2016). Identification of conserved and potentially regulatory small RNAs in heterocystous cyanobacteria. Front Microbiol 7: 48.
Klähn S, Schaal C, Georg J, Baumgartner D, Knippen G, Hagemann M, Muro-Pastor AM, Hess WR. (2015). The sRNA NsiR4 is involved in nitrogen assimilation control in cyanobacteria by targeting glutamine synthetase inactivating factor IF7. Proc Natl Acad Sci USA 112: E6243-6252.
Muro-Pastor AM. (2014). The heterocyst-specific NsiR1 small RNA is an early marker of cell differentiation in cyanobacterial filaments. mBio 5: e01079-14.
Muro-Pastor AM, Hess, WR. (2012). Heterocyst differentiation: from single mutants to global approaches. Trends Microbiol 20: 548-557.
Mitschke J, Vioque A, Haas F, Hess WR, Muro-Pastor AM. (2011). Dynamics of transcriptional start site selection during nitrogen stress-induced cell differentiation in Anabaena sp. PCC7120. Proc Nat Acad Sci USA 108: 20130-20135.
